Tecnología molecular para diferenciar cinco especies de Entamoeba
Por el equipo editorial de LabMedica en español
Actualizado el 23 Jun 2013
Se ha desarrollado un método de detección rápido, de alta eficiencia, para la detección y diferenciación simultánea de las especies de Entamoeba.Actualizado el 23 Jun 2013
Entre las especies de Entamoeba, sólo se considera a E. histolytica como patógena, causando enfermedades intestinales y extraintestinales, pero es morfológicamente idéntica a E. dispar y E. moshkovskii.
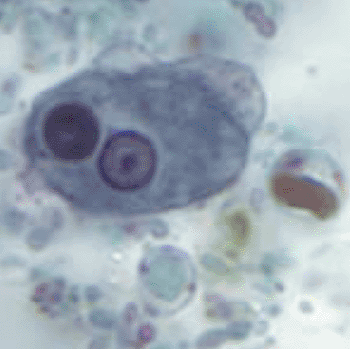
Imagen: Un trofozoito de Entamoeba histolytica (Fotografía cortesía de los Centros para el Control y Prevención de las Enfermedades).
Los científicos de la Universidad Federal de Río de Janeiro (Brasil) extrajeron 74 muestras de ADN de las heces humanas y nueve de estos extractos de ADN fueron de heces que eran positivas para las especies de Entamoeba por microscopía. Se realizó una amplificación mediante reacción en cadena de la polimerasa (PCR) con cebadores del gen del ácido ribonucleico ribosómico (rARN) 18S biotinilado de Entamoeba spp, diseñados para amplificar un fragmento que iba desde 382 hasta 429 pares de bases (pb) de las especies de Entamoeba estudiadas.
Todas las muestras parasitológicas positivas fueron analizadas mediante PCR en tiempo real y también se analizaron nueve muestras de heces por secuenciación del ADN. Además se seleccionaron las regiones de los fragmentos que podrían diferenciar entre E. histolytica, E. moshkovskii, E. dispar, E. hartmanii y E. coli para diseñar sondas de hibridación para unirlas a perlas Luminex (Austin, TX, EUA). El análisis fue estandarizado con muestras clonadas de ADN de cada especie y fue evaluado con 24 extractos de ADN a partir de muestras obtenidas de individuos diagnosticados con estas amebas en sus heces.
Los investigadores fueron capaces de identificar correctamente las cinco especies diferentes en todas las muestras estudiadas. De entre las 24 muestras analizadas por microscopía, la Secuenciación PCR/ADN y la PCR en tiempo real, tenían 100% de concordancia con el ensayo de PCR-Luminex para la identificación de E. dispar, E. moshkovskii, E. hartmanni, E. histolytica y E. coli. Ellos fueron capaces de identificar las muestras con más de una especie de Entamoeba mediante la realización de un ensayo Luminex y todas las cincuenta muestras de heces parasitológicamente negativas fueron negativas en el análisis.
Los autores concluyeron que el método podría ser utilizado en la detección de diagnóstico de especies de Entamoeba en muestras fecales. Esta prueba de diagnóstico fue útil para diferenciar claramente el patógeno E. histolytica de las otras especies y también para fortalecer los datos epidemiológicos sobre las especies de Entamoeba. El estudio fue publicado el 15 de marzo de 2013, en la revista Parasites & Vectors.
Enlaces relacionados:
The Federal University of Rio de Janeiro
Luminex