Desarrollan prueba novedosa para detectar e identificar patógenos
Por el equipo editorial de LabMedica en español
Actualizado el 05 Oct 2021
Los patógenos en entornos clínicos o de vida silvestre, como las muestras de sangre o saliva, por ejemplo, son particularmente difíciles de aislar, ya que pueden constituir fácilmente menos de una millonésima parte de una muestra, especialmente en las primeras etapas de una infección, cuando las concentraciones todavía son bajas y la detección es más crítica para los pacientes.Actualizado el 05 Oct 2021
Una amplia gama de esfuerzos de estudios metagenómicos se ven obstaculizados por el mismo desafío: bajas concentraciones de objetivos de interés combinadas con cantidades abrumadoras de señal de fondo. Aunque se puede utilizar PCR o captura de ADN sin tratamiento previo cuando hay una pequeña cantidad de organismos de interés, los desafíos de diseño se vuelven insostenibles para una gran cantidad de objetivos.
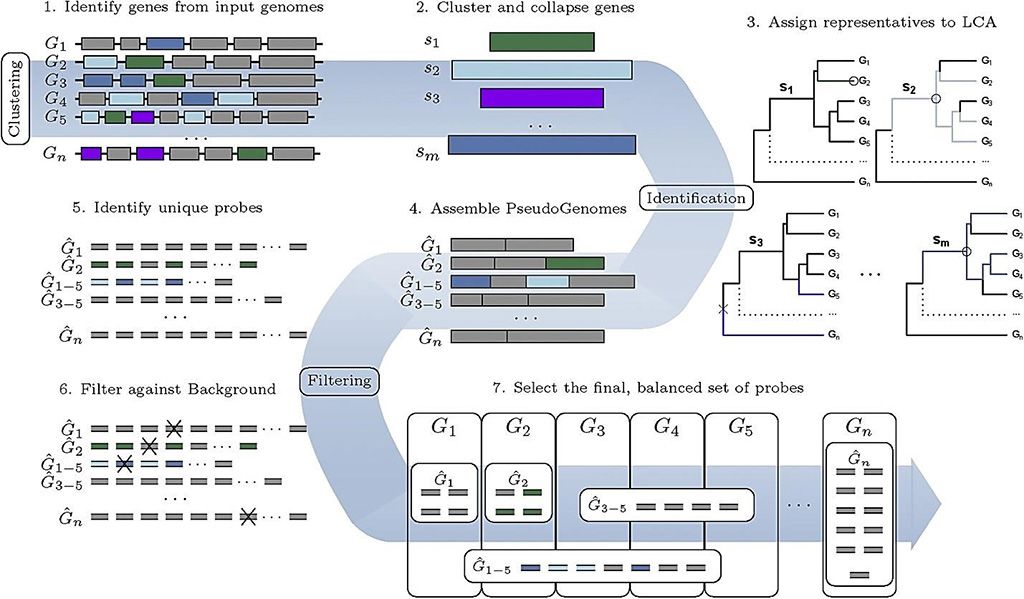
Imagen: El flujo de trabajo de la línea de productos en proyecto, HUBDesign: diseño de sonda para la captura simultánea y dirigida de diversos objetivos metagenómicos (Fotografía cortesía de la Universidad McMaster)
Un equipo de científicos de la Universidad McMaster (Hamilton, ON, Canadá) y sus colegas desarrollaron una herramienta sofisticada nueva que podría ayudar a proporcionar una alerta temprana de virus raros y desconocidos en el medio ambiente e identificar patógenos bacterianos potencialmente mortales que causan sepsis. El nuevo algoritmo es una herramienta avanzada que puede ayudar a desarrollar sondas para capturar trazas de patógenos, tanto conocidos como desconocidos provenientes de una amplia variedad de situaciones, como la transmisión de infecciones de animal a humano como el SARS-CoV-2 o los reservorios de monitoreo en el medio ambiente por posibles patógenos emergentes.
El equipo probó con éxito las sondas en toda la familia de coronavirus, incluido el SARS-CoV-2. Las sondas proporcionan un atajo al apuntar, aislar e identificar, las secuencias de ADN, de manera específica y simultánea, que se comparten entre organismos relacionados, con mayor frecuencia debido a la historia evolutiva o la ascendencia. Para demostrar las capacidades y la eficacia de la herramienta, el Diseño de Cebos Jerárquicos Únicos (The Hierarchical Unique Bait Design, HUBDesign), diseñaron y probaron dos conjuntos de sondas: un conjunto de sondas de coronavirus capaz de detectar simultáneamente todos los coronavirus secuenciados y un conjunto de sondas dirigidas a patógenos bacterianos asociados con la sepsis.
La mayoría (62,5%) de las sondas tienen objetivos específicos para un virus. De las sondas que se dirigen a varios virus, la mayoría (78,1%) se dirigen a dos o tres. Los tres conjuntos restantes de sondas se dirigen a loci específicos de merbecovirus y embecovirus (ambos son subgéneros de Betacoronavirus) y loci comunes al género Deltacoronavirus. Tanto el SARS-CoV-2 como el HCoV-NL63 tienen sondas en dos niveles en la jerarquía.
El conjunto de sondas HUBDesign para patógenos de la sepsis contenía 26.870 sondas dirigidas a patógenos bacterianos, que cubren el 2,09% de todos los nucleótidos en el conjunto de datos de entrada a una profundidad media de cobertura de 3,64x. Los investigadores demostraron la efectividad de las sondas para capturar la increíble variedad de patógenos asociados con la sepsis, una condición potencialmente mortal y de rápido desarrollo que ocurre cuando el cuerpo reacciona de forma exagerada a una infección que generalmente comienza en los pulmones, el tracto urinario, la piel o el tracto gastrointestinal.
Hendrik Poinar, PhD, profesor de Genética Evolutiva Molecular y autor principal del estudio, dijo: “Actualmente necesitamos formas más rápidas, económicas y concisas de detectar patógenos en muestras humanas y ambientales que democraticen la caza y esta línea de productos en proyecto hace exactamente eso”. El estudio fue publicado el 15 de septiembre de 2021 en la revista Cell Reports Methods.
Enlace relacionado:
Universidad McMaster