Usan método de amplificación isotérmica para detectar subtipos de Salmonella
|
Por el equipo editorial de LabMedica en español Actualizado el 24 Jun 2020 |

Imagen: Máquina de PCR en tiempo real, Corbett Rotor-Gene 6000 (Fotografía cortesía de Corbett Life Science).
La Salmonella es una causa común de enfermedades transmitidas por los alimentos en todo el mundo, incluida Australia. Más del 85% de los brotes de salmonelosis humana en Australia fueron causados por cinco serovares de Salmonella. La identificación rápida, exacta y sensible de los serotipos de Salmonella es vital para el diagnóstico y la vigilancia de la salud pública.
Los métodos tradicionales basados en cultivos para la detección de patógenos de Salmonella son lentos, laboriosos y costosos. Se han sugerido técnicas basadas en PCR (PCR y PCR en tiempo real) y técnicas de amplificación isotérmica, como la amplificación isotérmica mediada por bucle (LAMP), como alternativas. La amplificación de desplazamiento cruzado múltiple (MCDA) emplea 10 cebadores, en lugar de los seis usados en la LAMP o los dos que se usan en la PCR, para reconocer 10 regiones distintas, que mejoran su especificidad y sensibilidad.
Los biotecnólogos de la Universidad de Nueva Gales del Sur (Sídney, Australia) desarrollaron y evaluaron siete ensayos de MCDA para la detección rápida y la diferenciación de los cinco serovares de Salmonella más comunes en Australia: Typhimurium, Enteritidis, Virchow, Saintpaul e Infantis. Los conjuntos de cebadores para la MCDA se diseñaron dirigidos a siete marcadores genéticos específicos de los serovares/linajes, que fueron identificados mediante comparaciones genómicas. El equipo utilizó un total de 111 cepas.
Para evaluar los siete conjuntos de cebadores MCDA, se usó el ensayo invA MCDA con el gen específico de la especie de Salmonella como control positivo. Las reacciones MCDA se realizaron en una máquina de PCR en tiempo real, Corbett Rotor-Gene 6000 (Corbett Life Science, Sídney Australia) con el kit de amplificación de ADN, WarmStart LAMP (Nueva Inglaterra BioLabs, Melbourne, Australia) en un volumen total de 10 μL de mezcla de reacción incubada a 63°C durante 60 minutos y luego calentada a 95°C durante cinco minutos para detener la amplificación. Se usó la medición de colorante fluorescente LAMP en tiempo real para controlar la amplificación de MCDA cada minuto.
Los investigadores informaron que la sensibilidad y especificidad de los siete ensayos de MCDA se evaluaron utilizando 79 cepas objetivo y 32 cepas no objetivo. Todos los ensayos fueron altamente sensibles y específicos para los serovares objetivo, con una sensibilidad que oscilaba entre el 92,9% y el 100% y la especificidad entre el 93,3% y el 100%. El límite de detección de los siete ensayos de MCDA fue de 50 fg por reacción (10 copias) de ADN puro, y se detectaron resultados positivos en tan solo ocho minutos. Estos siete ensayos MCDA ofrecen un método de serotipificación rápido, exacto y sensible. Con una validación adicional en condiciones clínicamente relevantes, estos ensayos se podrían usar para la serotipificación independiente de cultivos de serovares comunes de Salmonella directamente de muestras clínicas.
Ruiting Lan, PhD, profesor y autor principal del estudio, dijo: “Reducir las infecciones de los fenotipos principales que causan la mayoría de los brotes, ayudará desde el nivel cero hasta el nivel clínico para reducir las infecciones por Salmonella”. El estudio fue publicado en la edición de mayo de 2020 de la revista The Journal of Molecular Diagnostics.
Enlace relacionado:
Universidad de Nueva Gales del Sur
Corbett Life Science
New England BioLabs
Los métodos tradicionales basados en cultivos para la detección de patógenos de Salmonella son lentos, laboriosos y costosos. Se han sugerido técnicas basadas en PCR (PCR y PCR en tiempo real) y técnicas de amplificación isotérmica, como la amplificación isotérmica mediada por bucle (LAMP), como alternativas. La amplificación de desplazamiento cruzado múltiple (MCDA) emplea 10 cebadores, en lugar de los seis usados en la LAMP o los dos que se usan en la PCR, para reconocer 10 regiones distintas, que mejoran su especificidad y sensibilidad.
Los biotecnólogos de la Universidad de Nueva Gales del Sur (Sídney, Australia) desarrollaron y evaluaron siete ensayos de MCDA para la detección rápida y la diferenciación de los cinco serovares de Salmonella más comunes en Australia: Typhimurium, Enteritidis, Virchow, Saintpaul e Infantis. Los conjuntos de cebadores para la MCDA se diseñaron dirigidos a siete marcadores genéticos específicos de los serovares/linajes, que fueron identificados mediante comparaciones genómicas. El equipo utilizó un total de 111 cepas.
Para evaluar los siete conjuntos de cebadores MCDA, se usó el ensayo invA MCDA con el gen específico de la especie de Salmonella como control positivo. Las reacciones MCDA se realizaron en una máquina de PCR en tiempo real, Corbett Rotor-Gene 6000 (Corbett Life Science, Sídney Australia) con el kit de amplificación de ADN, WarmStart LAMP (Nueva Inglaterra BioLabs, Melbourne, Australia) en un volumen total de 10 μL de mezcla de reacción incubada a 63°C durante 60 minutos y luego calentada a 95°C durante cinco minutos para detener la amplificación. Se usó la medición de colorante fluorescente LAMP en tiempo real para controlar la amplificación de MCDA cada minuto.
Los investigadores informaron que la sensibilidad y especificidad de los siete ensayos de MCDA se evaluaron utilizando 79 cepas objetivo y 32 cepas no objetivo. Todos los ensayos fueron altamente sensibles y específicos para los serovares objetivo, con una sensibilidad que oscilaba entre el 92,9% y el 100% y la especificidad entre el 93,3% y el 100%. El límite de detección de los siete ensayos de MCDA fue de 50 fg por reacción (10 copias) de ADN puro, y se detectaron resultados positivos en tan solo ocho minutos. Estos siete ensayos MCDA ofrecen un método de serotipificación rápido, exacto y sensible. Con una validación adicional en condiciones clínicamente relevantes, estos ensayos se podrían usar para la serotipificación independiente de cultivos de serovares comunes de Salmonella directamente de muestras clínicas.
Ruiting Lan, PhD, profesor y autor principal del estudio, dijo: “Reducir las infecciones de los fenotipos principales que causan la mayoría de los brotes, ayudará desde el nivel cero hasta el nivel clínico para reducir las infecciones por Salmonella”. El estudio fue publicado en la edición de mayo de 2020 de la revista The Journal of Molecular Diagnostics.
Enlace relacionado:
Universidad de Nueva Gales del Sur
Corbett Life Science
New England BioLabs
Últimas Diagnóstico Molecular noticias
- Marcador genético evita quimioterapia innecesaria a niños con leucemia de células T
- Prueba sanguínea de cuatro genes descarta infección pulmonar bacteriana
- Nueva prueba PCR mejora precisión diagnóstica de vaginosis bacteriana y vaginitis por Candida
- Nueva estrategia de edición de marcadores séricos mejora diagnóstico de enfermedades neurológicas
- Primera prueba genética de riesgo de diabetes tipo 1 permite detección temprana
- Análisis sanguíneo evita cirugías innecesarias en pacientes con cáncer gástrico de bajo riesgo
- Sistema automatizado pionero acelera diagnóstico del mieloma
- Perfiles de proteínas sanguíneas predicen riesgo de mortalidad por intervención médica temprana
- Primer análisis de sangre de su tipo detecta cáncer gástrico en pacientes asintomáticos
- Prueba molecular portátil detecta ITS en POC en 15 minutos
- Analizador de sobremesa ejecuta análisis químicos, inmunoensayos y hematología en un dispositivo
- Prueba POC de Bordetella ofrece resultados rápidos con precisión PCR
- Prueba sanguínea con punción detecta enfermedades 10 años antes de los síntomas
- Valores refinados de proteína C reactiva evaluan riesgo de sepsis en prematuros
- Análisis de sangre detecta con precisión patología amiloide cerebral en pacientes sintomáticos
- Nueva prueba molecular mejora precisión diagnóstica de enfermedad de Lyme
Canales
Química Clínica
ver canal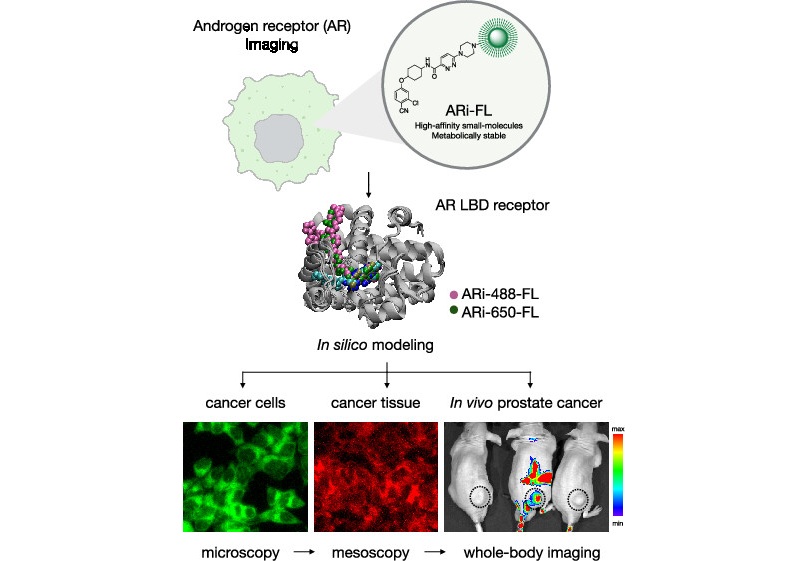
Sonda de imágenes químicas podría rastrear y tratar cáncer de próstata
El cáncer de próstata sigue siendo una de las principales causas de enfermedad y muerte en hombres, y muchos pacientes desarrollan resistencia a las terapias hormonales bloqueantes convencionales.... Más
Discrepancia entre dos pruebas comunes de función renal indica problemas de salud graves
La creatinina ha sido durante mucho tiempo el método estándar para medir la filtración renal, mientras que la cistatina C, una proteína producida por todas las células humanas, se ha recomendado como marcador... MásDiagnóstico Molecular
ver canal
Marcador genético evita quimioterapia innecesaria a niños con leucemia de células T
En el Reino Unido, aproximadamente 400 niños son diagnosticados con leucemia linfoblástica aguda (LLA) cada año, y aproximadamente el 15 % presenta un subtipo de LLA-T más agresivo. Si bien el enfoque... Más
Prueba sanguínea de cuatro genes descarta infección pulmonar bacteriana
Las infecciones de las vías respiratorias inferiores (IVRI) se encuentran entre las causas más comunes de prescripción de antibióticos; sin embargo, distinguir las infecciones bacterianas de las virales... Más
Nueva prueba PCR mejora precisión diagnóstica de vaginosis bacteriana y vaginitis por Candida
La vaginosis bacteriana (VB) afecta aproximadamente al 25 % de las mujeres en edad reproductiva, mientras que hasta el 75 % de las mujeres experimentan vaginitis por cándida (VC) al menos una vez en la vida.... MásHematología
ver canal
Análisis sanguíneo de actividad plaquetaria en mediana edad podría identificar riesgo temprano de Alzheimer
La detección temprana de la enfermedad de Alzheimer sigue siendo una de las mayores necesidades insatisfechas en neurología, sobre todo porque los cambios biológicos que subyacen al... Más
Medición de microvesículas podría detectar lesiones vasculares en pacientes con anemia falciforme
Evaluar la gravedad de la enfermedad de células falciformes (ECF) sigue siendo un reto, sobre todo al intentar predecir la hemólisis, el daño vascular y el riesgo de complicaciones... MásInmunología
ver canal
Nueva prueba distingue falsos positivos inducidos por vacuna de infección activa por VIH
Desde que se identificó el VIH en 1983, más de 91 millones de personas han contraído el virus y más de 44 millones han fallecido por causas relacionadas. Hoy en día, casi 40 millones de personas en todo... Más
Prueba de firma genética predice respuesta a tratamientos clave para cáncer de mama
Los inhibidores de DK4/6, combinados con terapia hormonal, se han convertido en un tratamiento fundamental para el cáncer de mama avanzado HR+/HER2–, ya que ralentizan el crecimiento tumoral... MásPatología
ver canalHerramienta de IA mejora a medicos detectando anomalías en células sanguíneas
El diagnóstico de trastornos sanguíneos depende del reconocimiento de anomalías sutiles en el tamaño, la forma y la estructura celular. Sin embargo, este proceso es lento, subjetivo y requiere años de... Más
Herramienta de IA analiza rápidamente imágenes complejas de cáncer para tratamiento personalizado
Las imágenes complejas de biopsia digital, que normalmente requieren hasta 20 minutos para ser evaluadas por un patólogo experto, ahora pueden analizarse en aproximadamente un minuto mediante una nueva... MásTecnología
ver canal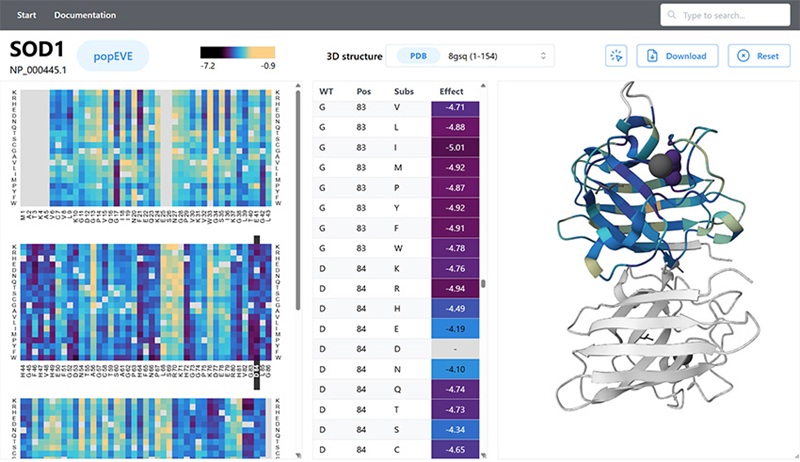
Modelo de inteligencia artificial podría acelerar diagnóstico de enfermedades raras
Identificar qué variantes genéticas causan enfermedades sigue siendo uno de los mayores desafíos de la medicina genómica. Cada persona porta decenas de miles de cambios en el... Más
Sensor de saliva con IA permite detección precoz del cáncer de cabeza y cuello
La detección precoz del cáncer de cabeza y cuello sigue siendo difícil porque la enfermedad produce pocos o ningún síntoma en sus primeras etapas, y las lesiones a menudo... MásIndustria
ver canal
Abbott adquiere Exact Sciences, empresa de detección de cáncer
Abbott (Abbott Park, IL, EUA) ha firmado un acuerdo definitivo para adquirir Exact Sciences (Madison, WI, EUA), lo que le permitirá entrar y liderar en segmentos de diagnóstico de cáncer... Más