Sistema rápido, ultrasensible, identifica las bacterias multi resistentes
Por el equipo editorial de LabMedica en español
Actualizado el 22 Dec 2020
La aparición mundial de bacterias multirresistentes (MDR) se asocia con importantes costos de morbilidad, mortalidad y atención médica. Los métodos de diagnóstico rápidos y exactos para detectar la resistencia a los antibióticos son fundamentales para la administración de antibióticos y las medidas de control de infecciones.Actualizado el 22 Dec 2020
Los métodos tradicionales para detectar la resistencia se basan en cultivar bacterias y probar su sensibilidad a un espectro de antibióticos. Estos métodos tardan de 48 a 72 horas en producir resultados y algunas cepas de bacterias no son fáciles de cultivar. Las pruebas biológicas moleculares son mucho más rápidas, pero incluso este método no ofrece resultados satisfactorios para todas las bacterias.
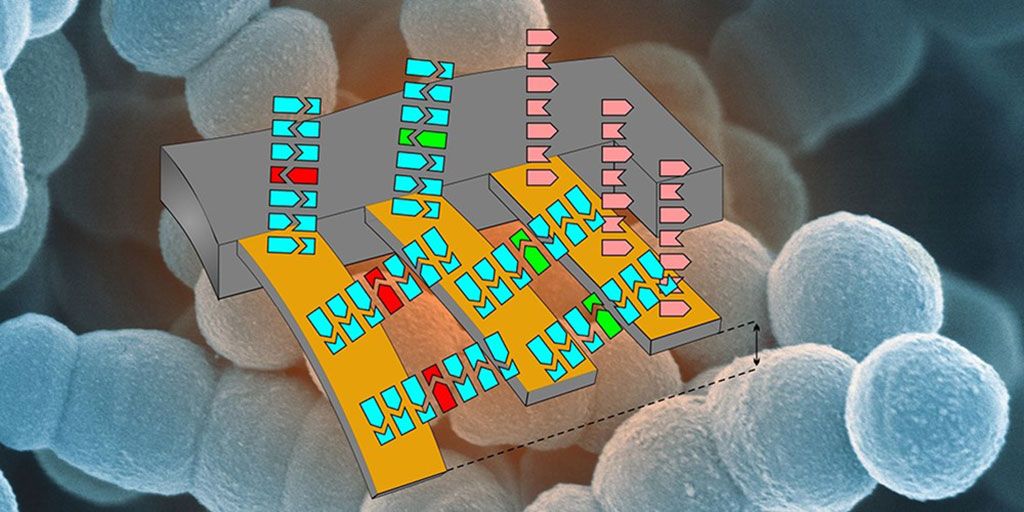
Imagen: Ilustración esquemática de la matriz en voladizo para detectar resistencia a los antibióticos. (Imagen: Departamento de Física y Laboratorio de Nanoimagenología, SNI, Universidad de Basilea).
Un equipo de científicos del Instituto de Nanociencias de Suiza (SNI, Basilea, Suiza) desarrolló un sistema de análisis en voladizo que les permitió detectar el ARN de una sola bacteria resistente a los antibióticos. Los aislados bacterianos, Pseudomonas aeruginosa y Enterococcus faecium, utilizados, provenían del biobanco de la división de Bacteriología Clínica y Micología del Hospital Universitario de Basilea (Basilea, Suiza). Se controló la calidad del ARN extraído utilizando Invitrogen Qubit 3.0 y NanoDrop 2000 (Thermo Fisher Scientific, Waltham, MA, EUA).
Con el nuevo sistema en voladizo, no es necesario amplificar o etiquetar las muestras para su análisis. El equipo comenzó uniendo secuencias de tres genes asociados con la resistencia a la vancomicina a los voladizos y luego expuso estos voladizos preparados a un flujo de ARN extraído de bacterias. Si las moléculas de ARN de los genes de resistencia estuvieran presentes, los fragmentos de ARN coincidentes se unirían a los voladizos, lo que haría que sufrieran una desviación a nanoescala que se podía detectar con un láser.
El equipo encontró una señal clara incluso con mutaciones puntuales. Este método permitió la detección no solo de genes de resistencia, sino también de mutaciones puntuales individuales asociadas con ellas. Para estudiar esto, los científicos utilizaron mutaciones puntuales acopladas a genes responsables de la resistencia a la ampicilina y otros antibióticos betalactámicos.
François Huber, Dr. Phil nat, profesor y primer autor del estudio, dijo: “La gran ventaja del método que hemos desarrollado es su velocidad y sensibilidad. Logramos detectar pequeñas cantidades de fragmentos de ARN específicos en cinco minutos”. En el caso de mutaciones únicas, las cantidades de ARN detectadas correspondieron a aproximadamente 10 bacterias. Cuando se trató de detectar genes de resistencia completos, los investigadores obtuvieron una señal clara incluso con una cantidad de ARN que correspondía a una sola bacteria. El estudio fue publicado el 30 de noviembre de 2020 en la revista Global Challenges.
Enlace relacionado:
Instituto de Nanociencias de Suiza
Hospital Universitario de Basilea