Sencillo método de microscopía óptica revela estructuras ocultas con un detalle asombroso
Actualizado el 20 Nov 2025
Comprender cómo se organizan las fibras microscópicas en los tejidos humanos es fundamental para revelar el funcionamiento de los órganos y cómo las enfermedades los alteran. Sin embargo, visualizar estas redes de fibras a nivel celular ha resultado complejo, lo que ha limitado la capacidad de los científicos para estudiar el deterioro estructural en afecciones como la esclerosis múltiple, la leucoencefalopatía y la enfermedad de Alzheimer. Un método de imagen recientemente desarrollado supera ahora esta barrera, ofreciendo una forma sencilla y económica de mapear la microestructura tisular con precisión micrométrica.
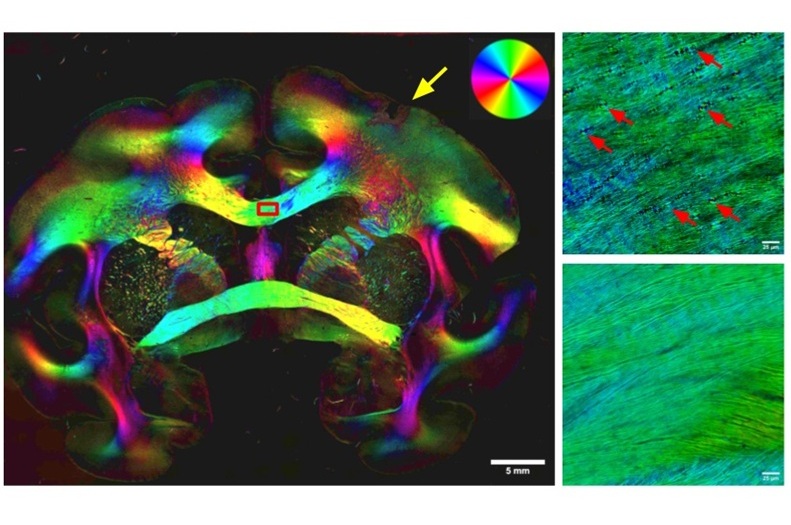
La tecnología, denominada imagen computacional de luz dispersa (ComSLI), ha sido desarrollada por investigadores de la Facultad de Medicina de la Universidad de Stanford (Stanford, California, EUA). ComSLI funciona aprovechando la forma en que la luz se dispersa de manera diferente según la orientación de las fibras microscópicas dentro del tejido. Una fuente de luz LED giratoria ilumina la muestra desde ángulos variables, mientras que una cámara de microscopio simple captura las sutiles diferencias de dispersión. Los algoritmos computacionales reconstruyen estos patrones en mapas de orientación de fibras con información sobre la microestructura: imágenes codificadas por colores que muestran la dirección, la densidad y la organización de las fibras en toda la muestra.
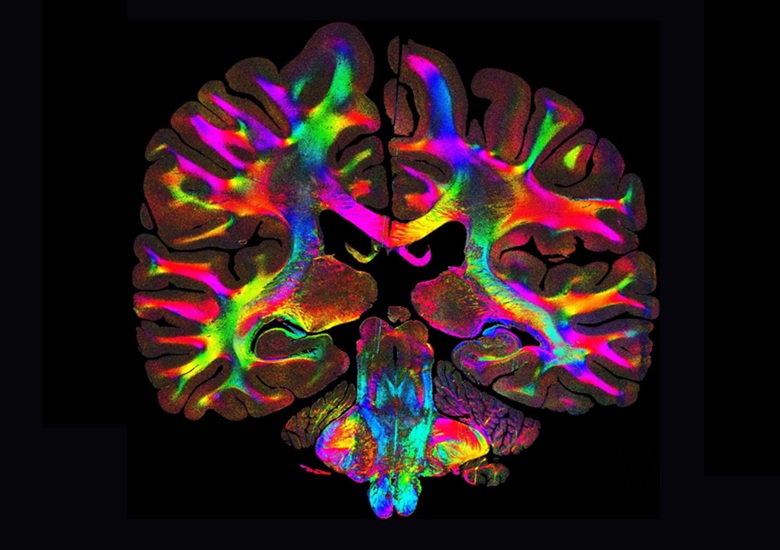
El sistema no requiere tinciones especiales, microscopios personalizados ni preparación especializada de portaobjetos. En estudios de validación, los investigadores demostraron que ComSLI funciona con prácticamente cualquier muestra histológica, incluyendo portaobjetos fijados en formalina e incluidos en parafina, comunes en laboratorios de patología de todo el mundo, así como tejidos frescos congelados, teñidos o sin teñir. La tecnología, presentada en Nature Communications, también reveló la arquitectura detallada de las fibras de una sección de cerebro humano preparada en 1904, demostrando que se pueden analizar muestras archivadas durante largos periodos sin que se degrade la información estructural.
Mediante ComSLI, el equipo visualizó intrincadas redes neuronales en secciones completas del cerebro humano. Mapearon cruces de fibras microscópicas en estructuras como el hipocampo y compararon tejido sano con muestras de pacientes con enfermedad de Alzheimer. En la muestra de Alzheimer, la vía perforante, una ruta crucial para las señales relacionadas con la memoria, era casi indetectable, y los cruces de fibras locales estaban significativamente reducidos. En contraste, el hipocampo sano presentaba fibras densas y ricamente interconectadas. Estos mapas microestructurales ofrecen una nueva perspectiva sobre cómo se deterioran los circuitos de la memoria en las enfermedades neurodegenerativas.
Además del cerebro, ComSLI logró obtener imágenes de tejidos musculares, óseos y vasculares. El músculo de la lengua mostró una disposición de fibras en capas que sustenta el movimiento; las muestras óseas revelaron colágeno alineado a lo largo de las vías de tensión mecánica; y las muestras arteriales mostraron capas alternas de colágeno y elastina responsables de la resistencia y la elasticidad. La capacidad de visualizar fibras en distintos órganos, especies y diapositivas archivadas durante décadas abre nuevas posibilidades en anatomía, patología, investigación evolutiva y mecanobiología.
"Esta es una herramienta que cualquier laboratorio puede usar. No se necesita preparación especializada ni equipos costosos", afirmó Michael Zeineh, MD, PhD, coautor principal. "Lo que más me entusiasma es que este enfoque abre la puerta a cualquiera, desde pequeños laboratorios de investigación hasta laboratorios de patología, para descubrir nuevos conocimientos a partir de las muestras que ya tienen".
Enlaces relacionados:
Facultad de Medicina de la Universidad de Stanford