Sensores de gas alimentados por ML detectan patógenos y RAM en POC
Actualizado el 25 Jul 2025
Un diagnóstico rápido y preciso es fundamental para mejorar la atención al paciente y combatir la amenaza global de la resistencia a los antimicrobianos (RAM). Los métodos tradicionales de diagnóstico de infecciones, como la cromatografía de gases con espectrometría de masas y la reacción de transferencia de protones con espectrometría de masas, si bien son eficaces, son costosos, técnicamente exigentes e inadecuados para la atención inmediata. La interpretación de los compuestos orgánicos volátiles (COV), las firmas químicas únicas emitidas por microbios y tejidos infectados, puede ser difícil debido a su complejidad y solapamiento. Además, estas señales de COV suelen estar influenciadas por variables ambientales, lo que aumenta la imprecisión diagnóstica. Un informe publicado en Cell Biomaterials explora el uso de sistemas de sensores combinados con modelos computacionales avanzados para detectar y clasificar COV microbianos con alta precisión.
Este informe, realizado por investigadores de la ETH Zurich (Zúrich, Suiza), presenta un enfoque prometedor que combina sensores de gas y aprendizaje automático para el diagnóstico de infecciones en tiempo real. El informe explora cómo los sensores de gas fabricados con óxidos metálicos nanoestructurados, polímeros conductores y compuestos híbridos podrían ofrecer una solución compacta, asequible y práctica para la detección de COV. Estos sensores miden los cambios en la resistencia eléctrica o la conductancia al exponerse a subproductos microbianos. Para decodificar los complejos patrones producidos por los COV, los investigadores evaluaron la viabilidad de integrar algoritmos de aprendizaje automático como máquinas de vectores de soporte (SVM), bosques aleatorios, redes neuronales de memoria a corto y largo plazo (LSTM) y potenciación de gradiente para clasificar los datos de los sensores y mejorar la precisión diagnóstica. Los modelos se evaluaron en cultivos bacterianos, tejidos infectados y biofluidos clínicos como orina y sangre, demostrando su capacidad para distinguir especies bacterianas y diferenciar entre cepas resistentes a los fármacos y susceptibles.
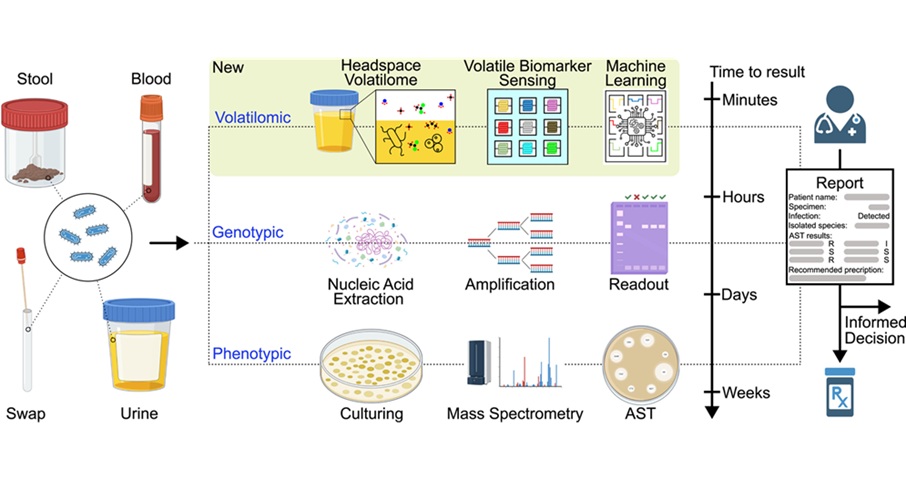
El equipo recopiló los hallazgos de estudios recientes que probaron estos sistemas de aprendizaje automático de sensores en cultivos bacterianos, muestras de tejido infectadas y biofluidos clínicos como orina y sangre. Los sistemas lograron alta sensibilidad y especificidad, identificando con precisión patógenos como Escherichia coli y Staphylococcus aureus, así como detectando perfiles de resistencia como los que involucran betalactamasas de espectro extendido. Los investigadores han enfatizado la necesidad de entrenar estos modelos de aprendizaje automático en conjuntos de datos completos que reflejen la variabilidad clínica para asegurar un rendimiento robusto. Los esfuerzos en curso incluyen la miniaturización de dispositivos para uso en el punto de atención, la funcionalización de superficies de sensores y la mitigación de interferencias ambientales como la humedad y la temperatura. Aunque se necesita mayor desarrollo y validación clínica, estos sistemas ofrecen un camino claro hacia diagnósticos rápidos y no invasivos que pueden complementar los métodos de laboratorio y respaldar una mejor administración de antimicrobianos.