Análisis de sangre identifica uso correcto de antibióticos
Por el equipo editorial de LabMedica en español
Actualizado el 16 Mar 2016
Actualizado el 16 Mar 2016
Las infecciones respiratorias agudas causadas por patógenos bacterianos o virales son algunas de las razones más comunes para la búsqueda de un carro médico y, a pesar de las mejoras en el diagnóstico basado en patógenos, la mayoría de los pacientes reciben antibióticos inapropiados.
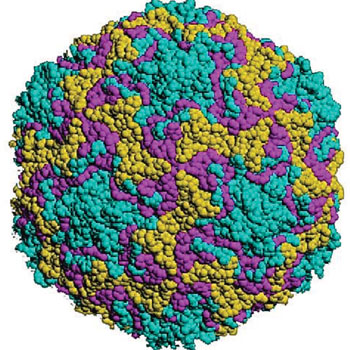
Imagen: Superficie molecular de la cápside del rinovirus humano, uno de los virus que causan infecciones respiratorias agudas (Fotografía cortesía de A. J. Cann).
El mal uso de los antibióticos es un factor clave para la resistencia a los antibióticos; si un paciente toma antibióticos para una infección viral, el medicamento seguirá atacando a las bacterias en el cuerpo, pero va a atacar a las bacterias saludables o beneficiosas. Esto puede generar propiedades de resistencia a los antibióticos que se pueden transmitir a otras bacterias.
Los científicos de la Universidad de Duke (Durham, Carolina del Norte, EUA) y sus colegas investigaron si los biomarcadores de respuesta del huésped ofrecen una aproximación diagnóstica alternativa para dirigir el uso de antimicrobianos. El estudio de cohorte observacional determina si los patrones de expresión de genes del huésped pueden discriminar entre la enfermedad no infecciosa y la enfermedad infecciosa bacteriana y entre las causas virales de infección respiratoria aguda en el ámbito de la atención aguda. Se midió la expresión de los genes en la sangre total periférica de 273 individuos con infección respiratoria aguda (IRA), de inicio comunitario o enfermedad no infecciosa, así como de 44 controles sanos, utilizando microarrays.
Mediante la medición de perfiles de expresión génica de los pacientes a partir de muestras de sangre, el equipo encontró que podían usar las firmas de genes previamente identificadas para identificar correctamente a los pacientes con virus de influenza, rinovirus, varios tipos de estreptococos y otras infecciones comunes, con una exactitud del 87%. Esta exactitud global de 238 de 273 individuos era concordante con la adjudicación clínica, que fue más exacta que la procalcitonina (78%), y tres clasificadores publicados de la infección bacteriana, versus la viral (78 a 83%). Los clasificadores desarrollados fueron validados externamente en cinco conjuntos de datos disponibles públicamente. Un sexto conjunto de datos a disposición del público incluyó a 25 pacientes con co-identificación de patógenos bacterianos y virales.
Los autores concluyeron que mediante la aplicación de los clasificadores de IRA podían definir cuatro grupos claros: una respuesta del huésped a la IRA bacteriana, la IRA viral, la coinfección, y una respuesta que no era bacteriana ni viral. Estos resultados crean una oportunidad para desarrollar y utilizar los clasificadores de la expresión de genes del huésped como plataformas de diagnóstico para combatir el uso inadecuado de antibióticos y la aparición de cepas resistentes a los antibióticos. Señalan que existe una limitación importante, dado que en la actualidad, se demora alrededor de 10 horas evaluar el perfil de expresión de genes de un paciente, pero el equipo dice que están en el proceso de trabajar con los desarrolladores para crear una prueba de una hora que pueda ser utilizada por los clínicos.
Christopher Woods, MD, MPH, un profesor de medicina y autor principal del estudio, dijo: “El escenario ideal, en caso de que esta prueba en última instancia, fuese aprobada para uso amplio, es que usted iría a la consulta del médico y recibiría sus resultados en el momento en que se reúne con su proveedor. Estamos trabajando para desarrollar una prueba que se pueda procesar en la mayoría de los laboratorios clínicos con los equipos existentes. Creemos que esto podría tener un impacto real sobre el uso adecuado de los antibióticos y orientar el uso de tratamientos antivirales en el futuro”. El estudio fue publicado el 20 de enero de 2016 en la revista Science Translational Medicine.
Enlace relacionado: