Validación analítica y uso clínico del análisis integral de perfiles genómicos
Por el equipo editorial de LabMedica en español
Actualizado el 09 Jun 2021
El cáncer tiene una gran carga clínica y las terapias oncológicas son caras. Se estima que se diagnosticarán 1.898.160 nuevos casos de cáncer y se prevé que ocurran más de 608.570 muertes en los EUA en 2021. Se espera que la prevalencia del cáncer aumente con el tiempo, lo que aumentará la necesidad insatisfecha de pruebas genómicas para ayudar a los médicos a tratar a los pacientes de una manera más precisa.Actualizado el 09 Jun 2021
La identificación de alteraciones genómicas mediante la secuenciación de próxima generación (NGS) se ha convertido en una herramienta clínica eficaz, especialmente para la oncología, ya que los marcadores moleculares pueden orientar el tratamiento personalizado. Las opciones disponibles actualmente para el perfil de tumores incluyen inmunohistoquímica (IHC), hibridación in situ fluorescente (FISH) y, más recientemente, secuenciación de próxima generación de panel pequeño (NGS).
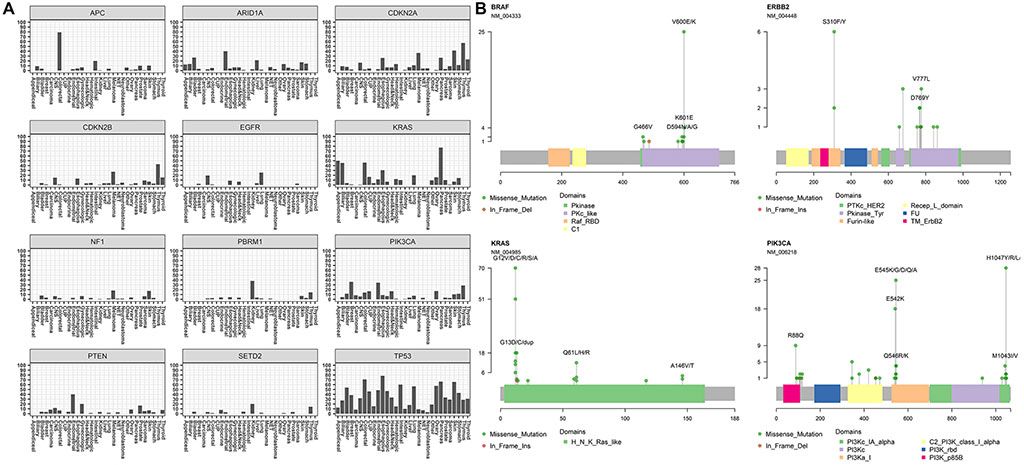
Imagen: Perfiles de mutación de genes clínicamente accionables en GEM ExTra. Los doce genes más reportados y su distribución de mutaciones entre los tipos de tumores (Fotografía cortesía de Ashion Analytics)
Los científicos de Ashion Analytics, LLC (Phoenix, AZ, EUA), desarrollaron y validaron analíticamente su ensayo integral de perfil genómico, GEM ExTra. El ensayo es para pacientes con tumores sólidos avanzados que utilizan la secuenciación de próxima generación para caracterizar exomas completos empleando una metodología de sustracción normal de tumor pareado. La prueba se utilizó en más de 1.400 muestras de pacientes durante un período de abril de 2018 y diciembre de 2019 en todos los centros de cáncer para detectar múltiples alteraciones procesables en una variedad de tipos de cáncer.
El ensayo detecta variantes de un solo nucleótido (SNV), indeles, alteraciones del número de copias focales (CNA), región promotora TERT, así como la carga de mutaciones tumorales (TMB) y el estado de inestabilidad de microsatélites (MSI). Además, el ensayo incorpora la secuenciación del transcriptoma completo de la muestra del tumor que permite la detección de fusiones de genes y selecciona transcripciones especiales, incluidos los eventos de omisión del exón 14 de AR-V7, EGFR vIII, EGFRvIV y MET. El ensayo tiene una cobertura diana media de 180 × para el ADN normal (línea germinal) y 400 × para el ADN tumoral, incluido un diseño de sonda mejorado para facilitar la secuenciación de regiones difíciles.
El equipo descubrió que el 83,9% de las muestras de tumores albergaban, al menos, una alteración clínicamente procesable (definida como positiva) y el resto definida como negativa, con un total de 1.267 informes positivos y 242 negativos (Tasa de detección: 2018 = 76,4%, 2019 = 86,4%). En general, se identificaron 3.535 mutaciones clínicamente accionables en la cohorte (1.864 mutaciones únicas), con una mediana de dos alteraciones clínicamente accionables por tumor (media = 2,93 ± 2,37) que muestran una amplia variación entre los tipos de cáncer. Los tumores con el mayor número de mutaciones accionables incluyeron piel (4,9 ± 2,2), endometrio (4,5 ± 3,9) y colorrectal (4,1 ± 3,5). Estos resultados generalmente concuerdan con estimaciones previas de eventos impulsores por paciente en estos tipos de tumores. Esto es algo más bajo de lo que se informó anteriormente en un estudio pan-cáncer (4,6/tumor) de genomas completos, que también incluyó alteraciones en el número de copias del controlador que no se consideraron procesables con GEM ExTra. La media de SNV codificantes (es decir, codón de terminación sin sentido, sin sentido) fue de 1,9 ± 1,4 por tumor, que se encuentra dentro del rango de mutaciones impulsoras predichas en el cáncer.
Los científicos concluyeron que habían desarrollado y validado analíticamente un ensayo de perfil genómico completo con un tiempo de respuesta de 14 días que se puede adaptar a todas las necesidades futuras de análisis de tumores debido al análisis combinado de ADN y ARN. El ensayo GEM ExTra no solo utiliza WES para la elaboración de perfiles de ADN tumoral, sino que también identifica variantes de transcripción y genes de fusión clínicamente procesables a través de la secuenciación de ARN, lo que garantiza que GEM ExTra será integral en el futuro. El estudio fue publicado el 13 de abril de 2021 en la revista Oncotarget.
Enlace relacionado:
Ashion Analytics