La secuenciación del genoma completo ayuda al diagnóstico de enfermedades raras
Por el equipo editorial de LabMedica en español
Actualizado el 13 Apr 2021
Los diagnósticos de enfermedades genéticas están en revolución, debido a los descubrimientos en la tecnología de secuenciación y de análisis de datos. El potencial para transformar la medicina clínica usando la genómica es alto, especialmente dentro del ámbito de las enfermedades raras. Actualizado el 13 Apr 2021
Las enfermedades raras constituyen un grupo grande y heterogéneo de diagnósticos que incluyen más de 8.000 condiciones distintas de las cuales la gran mayoría tiene una base genética. Cada enfermedad individual es rara, pero cuando se consideran como un grupo, las enfermedades raras son comunes con una prevalencia total de aproximadamente de 6% a 8%.

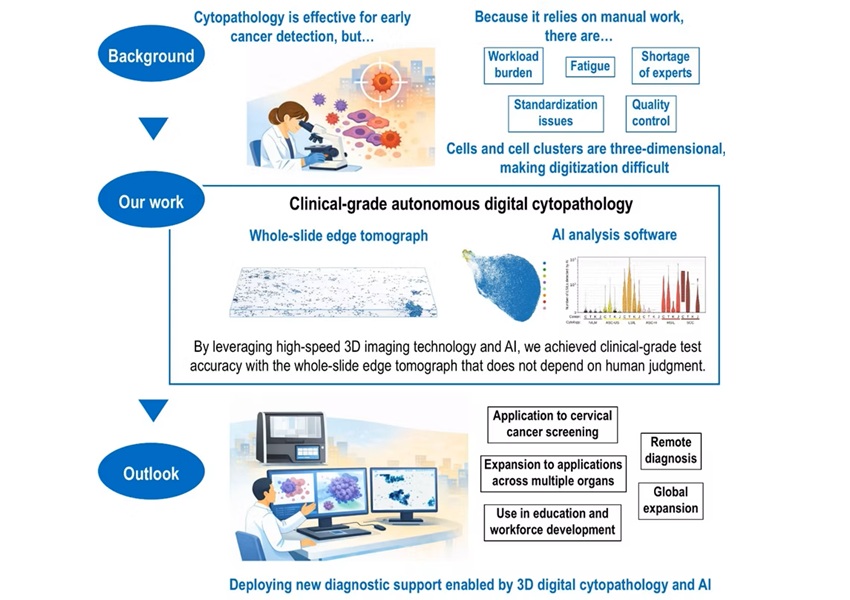
Imagen: El analizador MassARRAY Dx es un analizador genético multiplex de sobremesa que simplifica el entorno complejo de la genética clínica, con datos fáciles de interpretar, detección flexible de biomarcadores y desempeño robusto (Fotografía cortesía de Agena Biosciences)
Científicos moleculares médicos del Instituto Karolinska (Estocolmo, Suecia) y sus colegas, secuenciaron los genomas de 3.219 pacientes en un programa de cinco años. La secuenciación clínica del genoma completo (SGC) fue implementada gradualmente en el Centro de Medicina Genómica Karolinska-Enfermedades Raras (GMCK-RD), en el transcurso de cinco años. Al inicio la secuenciación fue realizada en el HiSeq X Ten, pero se cambió al NovaSeq 6000 (Illumina, San Diego, CA, EUA) en diciembre de 2018.
Generalmente, las muestras fueron analizadas de manera escalonada, en las que primero se analizaron para variantes asociadas con la presunta enfermedad del paciente, algunas veces con paneles muy grandes de genes de aproximadamente 4.000 genes, ante de ser escaladas para análisis adicional o análisis de genoma completo, si no se hizo un diagnóstico. Para asegurar que no hay mezcla de la muestra durante el procesamiento de SGC, una alícuota del ADN extraído fue genotipificada para 51 SNPs usando tecnología MassARRAY (Agena Biosciences, San Diego, CA, EUA).
Los científicos descubrieron variantes en 754 genes de enfermedades diferentes, con la mayoría afectando comúnmente los genes COL2A1 y FKRP. Varias variantes también fueron recurrentes, algunas de las cuales eran mutaciones fundadoras conocidas, como Leu27Ile en FKRP, que fueron homocigotas en 12 personas con distrofia muscular de la cintura de las extremidades, y expansiones homocigotas en el gen RFC1 entre personas con ataxia cerebelosa, neuropatía, y síndrome de arreflexia vestibular. Sin embargo, otros pacientes tenían variantes que se sabe surgen recurrentemente como mutaciones de novo, como una que afecta el gen PRRT2 que causa convulsiones.
Para algunos pacientes tener un diagnóstico molecular puede cambiar su tratamiento. Como señaló el equipo, pacientes con errores innatos del metabolismo de inicio agudo con frecuencia se tratan con infusión de glucosa, pero para los pacientes con deficiencia de piruvato deshidrogenasa, esto puede ser peligroso. En cambio, esos pacientes son tratados con una dieta cetogénica. Los científicos generaron diagnósticos moleculares para 1.287 pacientes, o 40%, con un tiempo medio de entrega de resultados de 13 días.
Anna Wedell, MD, PhD, una profesora de medicina molecular y cirugía y una coautora senior del estudio, dijo: “La mayoría de los pacientes no se habrían sometido a investigaciones genéticas con los métodos convencionales. Hemos integrado WGS profundamente en la clínica, haciendo que la genómica esté disponible en escenarios completamente nuevos incluyendo las situaciones médicas agudas para una amplia gama de grupos de enfermedades”. El estudio fue publicado el 17 de marzo de 2021 en la revista “Genome Medicine”.
Enlace relacionado:
Instituto Karolinska
Illumina
Agena Biosciences