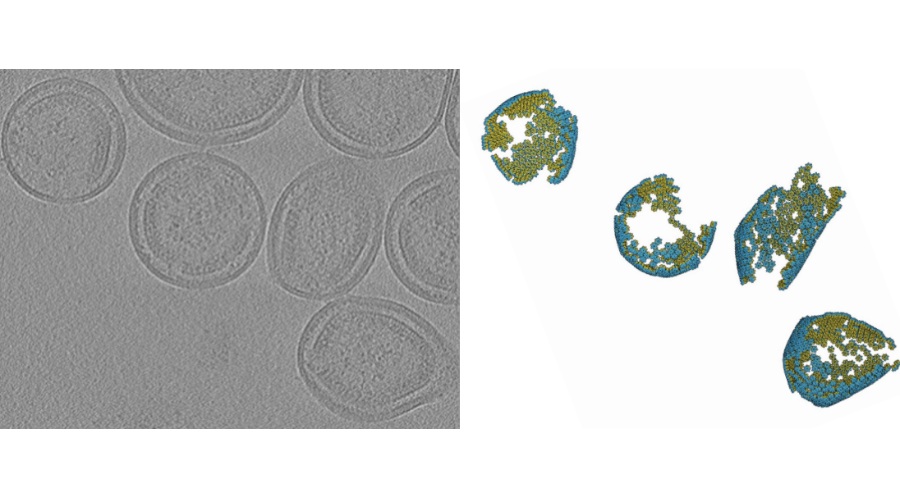
Asocian bacteria intestinal específica al síndrome de intestino irritable
Por el equipo editorial de LabMedica en español
Actualizado el 08 Dec 2020
La incidencia del síndrome del intestino irritable (SII) aumenta abruptamente después de un episodio de gastroenteritis, lo que sugiere un posible papel causal de la perturbación microbiana. Los estudios de composición de la microbiota intestinal se basan mayoritariamente en heces.Actualizado el 08 Dec 2020
Las bacterias asociadas al moco y las heces representan poblaciones distintivas, y es más probable que estas últimas influyan en el epitelio. En particular, la presencia de bacterias en la capa interna de moco, podría resultar en estrés epitelial y activación inmune. Los análisis de la microbiota fecal no han demostrado alteraciones consistentes en el SII.
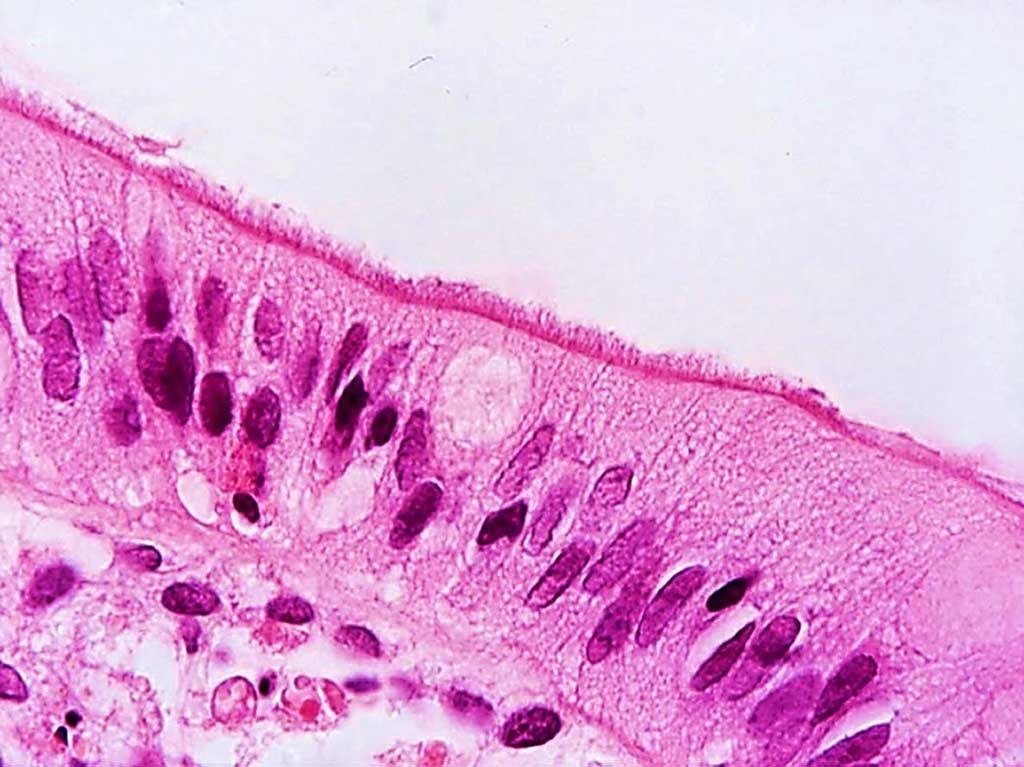
Imagen: Aspecto de un “borde en cepillo falso” de células de Brachyspira pilosicoli unidas por un extremo celular a la superficie luminal de los enterocitos del colon humano en un paciente diagnosticado con espiroquetosis intestinal humana (Fotografía cortesía de David J. Hampson, PhD, DSc).
Los científicos biomédicos de la Universidad de Gotemburgo (Gotemburgo, Suecia) y sus colegas, incluyeron de forma prospectiva a 62 pacientes con SII y 31 controles normales a quienes les realizaron una sigmoidoscopia con muestreo de biopsias en metanol-Carnoy para histología/inmunohistoquímica futura y análisis de PCR en tiempo real. En un subconjunto de participantes seleccionado al azar (la primera cohorte exploratoria, SII n = 22, sano n = 14), se recogió moco de biopsias de colon sigmoide ex vivo y se analizó mediante espectrometría de masas (EM).
Se prepararon muestras de moco para la EM de acuerdo con una versión modificada del protocolo de preparación de muestras con ayuda de filtro (FASP). La cromatografía de nano-líquidos en tándem con la EM se realizó en un instrumento Q-Exactive (Thermo Fisher Scientific, Bremen, Alemania). Se realizó histología e inmunohistoquímica en cortes de tejido. Las secciones se examinaron usando un microscopio epifluorescente Eclipse E-1000 (Nikon, Tokio, Japón). Todas las reacciones de PCR se procesaron por triplicado, utilizando un sistema en tiempo real Bio-Rad CFX96 (Bio-Rad, Hércules, CA, EUA).
Los investigadores informaron que el análisis metaproteómico de las muestras de moco del colon identificaba péptidos de especies de Brachyspira potencialmente patógenas en un subconjunto de pacientes con SII. Utilizando múltiples métodos de diagnóstico, se detectó colonización de la mucosa por Brachyspira en un total de 19/62 (31%) pacientes con SII de dos cohortes prospectivas, frente a 0/31 voluntarios sanos. La prevalencia de colonización por Brachyspira en el SII con diarrea (SII-D) fue del 40% en ambas cohortes. La unión de Brachyspira a la membrana apical de los colonocitos se observó en el 20% de los pacientes con SII y se asoció con tránsito oroanal acelerado, inflamación leve de la mucosa, activación de mastocitos y alteraciones de las vías moleculares vinculadas a la captación bacteriana y la homeostasis iónica-fluida. Según la discriminación de especies por PCR en tiempo real, el 50% de los pacientes con espiroquetasis fueron colonizados por B. pilosicoli; otros tenían B. aalborgi o la especie, estrechamente relacionada sin confirmación, B. hominis.
Magnus Simrén, MD, PhD, profesor de gastroenterología y coautor del estudio, dijo: “El estudio sugiere que la bacteria se puede encontrar en aproximadamente un tercio de las personas con SII. Queremos ver si esto se puede confirmar en un estudio más amplio, y también vamos a investigar si Brachyspira causa síntomas en el SII y cómo lo hace. Nuestros hallazgos pueden abrir oportunidades completamente nuevas para tratar y quizás incluso curar a algunos pacientes con SII, especialmente aquellos que tienen diarrea”.
Los autores concluyeron que la colonización de la mucosa por Brachyspira fue significativamente más común en el SII y se asoció con características clínicas, histológicas y moleculares claras. Las observaciones sugieren un papel para Brachyspira en la patogénesis del SII, particularmente el SII-D. El estudio fue publicado el 11 de noviembre de 2020 en la revista GUT.
Enlace relacionado:
Universidad de Gotemburgo
Thermo Fisher Scientific
Nikon