Firmas del análisis de una sola célula se asocian con los resultados del ependimoma pediátrico
Por el equipo editorial de LabMedica en español
Actualizado el 29 Jul 2020
Un ependimoma es un tumor que surge del epéndima, un tejido del sistema nervioso central. Por lo general, en casos pediátricos, la ubicación es intracraneal, mientras que en adultos es espinal. La ubicación común de los ependimomas intracraneales es el cuarto ventrículo.Actualizado el 29 Jul 2020
Los ependimomas constituyen aproximadamente el 5% de los gliomas intracraneales adultos y hasta el 10% de los tumores infantiles del sistema nervioso central (SNC). Su aparición parece alcanzar su punto máximo a los 5 años y luego nuevamente a los 35. Se desarrollan a partir de células que recubren las cavidades huecas del cerebro y el canal que contiene la médula espinal.

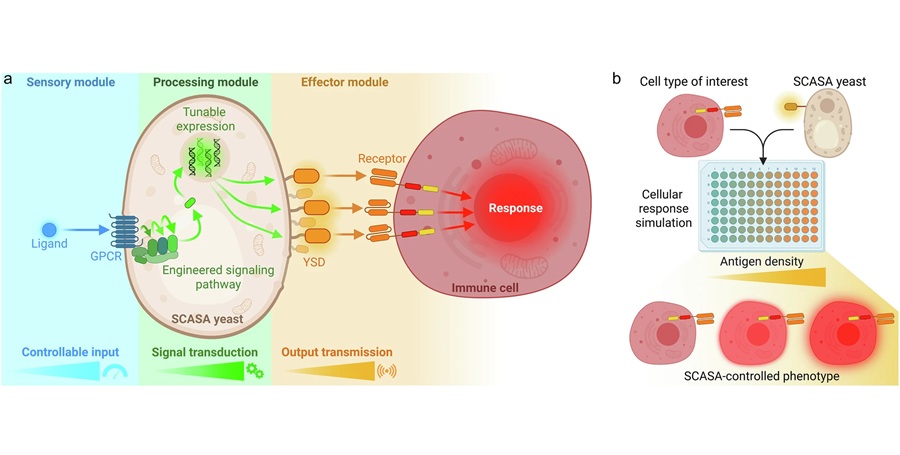
Imagen: El clasificador de células SH800S de sobremesa permite la clasificación de una amplia gama de tamaños celulares para muchas aplicaciones que utilizan chips de clasificación microfluídica de 70 μm, 100 μm y 130 μm (Fotografía cortesía de Sony Biotechnology).
Un equipo de oncólogos pediátricos con sede en el Centro de Cáncer Dana-Farber (Boston, MA, EUA) y sus colegas, generaron datos transcriptómicos de células individuales en 20 muestras de tumores quirúrgicos frescos de 18 pacientes, así como en ocho modelos celulares y dos xenoinjertos, derivados de los pacientes. Al mismo tiempo, realizaron una secuencia de ARN de un solo núcleo en 14 muestras de ependimoma congelado. En total, analizaron 74.927 células o núcleos tumorales únicos y, basándose en los patrones de metilación del ADN de las muestras, determinaron los subgrupos moleculares de los tumores.
El tejido tumoral se disoció mecánicamente, seguido de digestión enzimática a base de papaína durante 30 minutos a 37°C, con un kit de disociación de tumores cerebrales (Miltenyi Biotec, Bergisch Gladbach, Alemania). La clasificación de células individuales se realizó en un clasificador de células SH700 (Sony Biotechnology, San José, CA, EUA) usando láseres de 488 nm (calceína AM, filtro de emisión 530/30) y 633 nm (TO-PRO3, filtro de emisión 665/30). La amplificación completa del transcriptoma, la preparación de la biblioteca y la secuenciación de células y núcleos individuales, se realizaron siguiendo el protocolo modificado, Smart-seq2. Los núcleos individuales se procesaron mediante los kits de reactivos 10X Chromium Single Cell 3, basados en microfluídica v3 (10X Genomics, Pleasanton, CA, EUA).
Los investigadores informaron que los transcriptomas de las células que se encuentran dentro de los ependimomas eran similares a los de las células cerebrales normales. Sin embargo, notaron que las células tumorales parecían haberse detenido en varios puntos a lo largo del proceso de diferenciación. Dentro de los ependimomas, descubrieron tres trayectorias de diferenciación: células de tipo ependimario, de tipo progenitor glial y de tipo neuronal. En las muestras de ependimoma de fosa posterior, el grupo de fosa posterior A (PF-A) es el más agresivo de los subgrupos y el grupo de fosa posterior B (PF-B) se relaciona con un mejor pronóstico para los pacientes: descubrieron nueve metaprogramas transcripcionales recurrentes. Dos programas se vincularon con genes del ciclo celular y se encontraron particularmente entre las muestras de PF-A. Otros metaprogramas se asociaron con tipos de células maduras, astrocitos o células madre inmaduras y precursores de linaje neuronal o glial.
El equipo identificó 10 metaprogramas transcripcionales, incluidos dos asociados con genes del ciclo celular y otros vinculados a tipos de células radiales de tipo glial o precursor neuronal, entre ocho muestras de ependimoma supratentorial. Los investigadores encontraron que estados celulares indiferenciados también fueron más comunes entre los tumores con peor pronóstico, como los tumores PF-A típicamente más agresivos. Los grupos más benignos como PF-B o PF-subependimoma tenían poblaciones celulares menos proliferativas y más diferenciadas. Dentro de los datos de expresión de ARN en masa, las firmas transcripcionales de los tumores se correlacionaron con la supervivencia.
Los autores concluyeron que esta desconvolución de subpoblaciones heterogéneas (ependimoma) identifica firmas transcriptómicas malignas clave e identifica tumores de alto riesgo y posteriormente informa el desarrollo de tratamientos más efectivos contra el ependimoma. El estudio fue publicado el 13 de julio de 2020 en la revista Cancer Cell.
Enlace relacionado:
Centro de Cáncer Dana-Farber
Sony Biotechnology
10X Genomics