Clasifican un cáncer de mama triple negativo a partir de la secuenciación de todo el genoma
Por el equipo editorial de LabMedica en español
Actualizado el 23 Oct 2019
Un diagnóstico de cáncer de mama triple negativo significa que los tres tipos más comunes de receptores conocidos para alimentar la mayor parte del crecimiento del cáncer de seno: el estrógeno, la progesterona y el gen HER-2/neu están ausentes en el tumor canceroso.Actualizado el 23 Oct 2019
El cáncer de mama triple negativo (TNBC, por sus siglas en inglés) puede ser más agresivo y difícil de tratar. Además, es un cáncer que tienen mayor probabilidad de propagarse y recurrir. La etapa del cáncer de mama y el grado del tumor influirán en el pronóstico. La tasa de supervivencia a cinco años para los TNBC es de alrededor del 77% frente al 93% para los otros tipos de cáncer de mama.
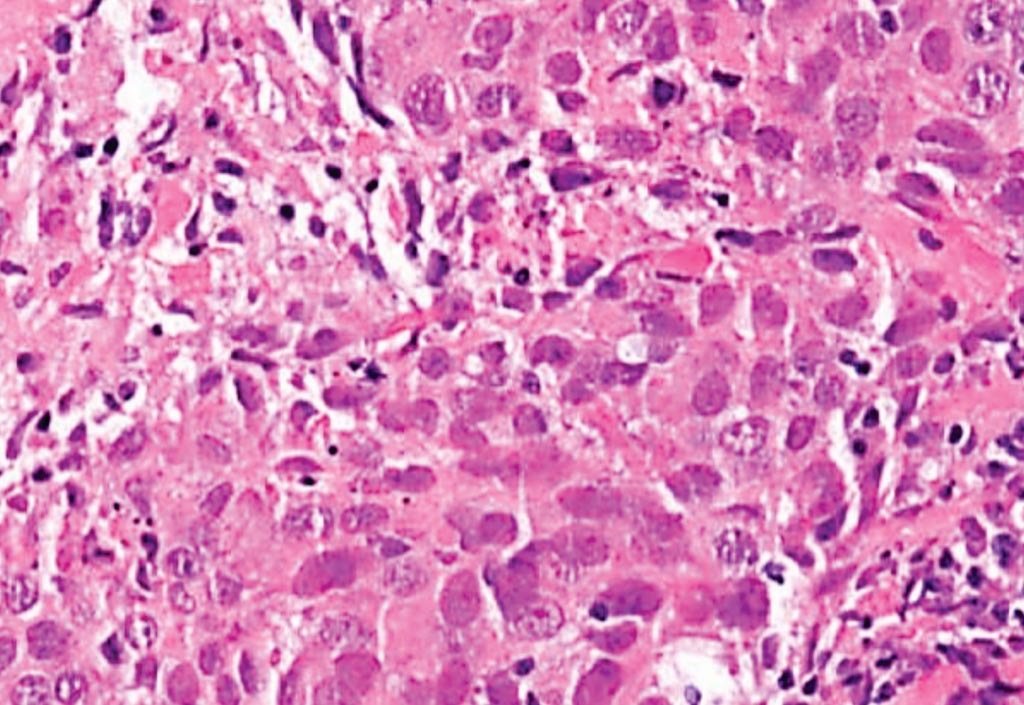
Imagen: Una histopatología del carcinoma ductal invasivo de mama de alto grado (no especificado de otra manera, grado 3). Este es un ejemplo de un cáncer de seno triple negativo, carcinoma tipo basal (Fotografía cortesía del Centro de Cáncer Lee Moffitt).
Un equipo internacional de científicos dirigido por aquellos en la Universidad de Lund (Lund, Suecia) secuenciaron 254 tumores TNBC de pacientes del proyecto de la Red de Análisis de Cáncer de Mama de Suecia (SCAN-B), un estudio observacional en curso basado en la población de pacientes con cáncer de mama en el sur de Suecia. Al aplicar el algoritmo de aprendizaje automático HRDetect, desarrollado previamente, a las secuencias del genoma, los investigadores dividieron los tumores en tres grupos: bajo, intermedio y alto, en función de cuán deficientes eran en la reparación génica basada en recombinación homóloga. Estos grupos tenían diferentes pronósticos y respuestas a los tratamientos.
Después de determinar los posibles impulsores somáticos y las mutaciones de la línea germinal patógena para esta cohorte, el equipo aplicó el algoritmo basado en la firma mutacional HRDetect para examinar la deficiencia de reparación de recombinación homóloga o de estas muestras. Casi el 60% de los tumores triple negativos se clasificaron como HRDetect-altos; alrededor del 35% eran HRDetect-bajo; y alrededor del 5% cayó en un grupo intermedio. Los científicos informaron que el grupo HRDetect-alto se enriqueció para pacientes menores de 50 años, con tumores de alto grado y un subtipo de expresión de tipo basal. Pero como señalaron, este grupo también incluyó tumores con coloración para los receptores de estrógeno, de pacientes de mediana edad, y con perfiles de expresión génica no basales, lo que sugiere que el fenotipo HRDetect-alto está enriquecido para los tumores basales típicos encontrados entre pacientes jóvenes con enfermedad triple negativa, pero también abarca a otros. Esto, argumentaron, sugiere que HRDetect representa un medio novedoso para estratificar tumores.
Los pacientes cuyos tumores cayeron en estos diferentes grupos de HRDetect también respondieron de manera diferente al tratamiento. Los pacientes en el grupo HRDetect-alto tuvieron los mejores resultados de los tres. Estos tumores tenían más probabilidades de responder a la quimioterapia adyuvante, así como a los inhibidores de PARP. Mientras tanto, el grupo intermedio HRDetect tuvo los peores resultados, aunque el grupo bajo HRDetect también tuvo malos resultados. Pero como señalaron los científicos, los perfiles moleculares de los grupos HRDetect-intermedio y bajo sugirieron posibles objetivos accionables. El grupo intermedio HRDetect, por ejemplo, se enriqueció para tumores con amplificaciones de ciclina E1 (CCNE1) y para hipermutadores de una firma de reordenamiento de duplicaciones largas en tándem. El grupo HRDetect-bajo, mientras tanto, se enriqueció por alteraciones en la vía PIK3CA/AKT1.
Johan Staaf, PhD, profesor asociado de oncología y patología, y primer autor del estudio, dijo: “Al utilizar la secuenciación del genoma completo, podemos discriminar realmente los tumores que pueden o no responder a los medicamentos actuales entre pacientes con cáncer de mama triple negativo, un tipo de cáncer de mama que todavía tenemos dificultad para tratar”. El estudio fue publicado el 30 de septiembre de 2019 en la revista Nature Medicine.
Enlace relacionado:
Universidad de Lund