El análisis del cáncer gástrico identifica cuatro subtipos
Por el equipo editorial de LabMedica en español
Actualizado el 06 Mar 2019
En estudios previos de cánceres gástricos (CG), los análisis genómicos y transcriptómicos han identificado firmas moleculares asociadas con los fenotipos de la enfermedad, como los subtipos de pacientes y la supervivencia. El nuevo análisis proteogenómico de los cánceres gástricos difusos (CG) en poblaciones jóvenes, identificó cuatro subtipos de la enfermedad.Actualizado el 06 Mar 2019
El Atlas del Genoma del Cáncer (TCGA, por sus siglas en inglés) identificó cuatro subtipos de CG y firmas moleculares asociadas: tumores positivos al virus de Epstein-Barr (EBV) con mutaciones recurrentes de PIK3CA, hipermetilación del ADN y amplificación de JAK2, CD274 y PDCD1LG2; tumores con inestabilidad de microsatélite con altas tasas de mutación; tumores genómicamente estables enriquecidos por variantes histológicas difusas y mutaciones en RHOA; y tumores con inestabilidad cromosómica mostrando aneuploidía y amplificaciones de genes que codifican receptores tirosina quinasas.
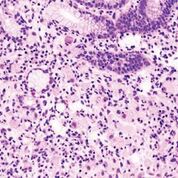
Imagen: Una histopatología del adenocarcinoma gástrico que representa una variante de células en anillo de sello identificada en una muestra de biopsia endoscópica (Fotografía cortesía del KGH).
Un gran equipo de científicos que colaboran con la Universidad de Corea (Seúl, República de Corea) recogieron tumores pareados y tejidos normales adyacentes, así como muestras de sangre, de 80 pacientes con CG de inicio temprano (CGIT) menores de 45 años. Estos 80 tumores incluían 74 tumores difusos, tres intestinales, dos tipos mixtos y un tumor mioblástico inflamatorio. Para cada paciente, el equipo realizó la secuenciación del exoma de los tumores y de las células mononucleares de sangre periférica, así como la secuenciación del mARN del tumor pareado y de los tejidos normales adyacentes.
El equipo utilizó datos de secuenciación del exoma e identificó 56.502 variantes de un solo nucleótido no sinónimas y 3.598 indels de cambio de marco. Además, encontraron 11.938 genes expresados en el tumor y muestras normales adyacentes, en promedio, en los datos del mARN. Luego utilizaron estas variantes y expresaron transcripciones de cada paciente para construir una base de datos específica de la muestra e identificaron 156.135 péptidos, 28.944 fosfopéptidos y 4.376 N-glicopéptidos de los proteomas globales, fosfoproteomas y N-glicoproteomas, respectivamente. Estos péptidos se asignaron a 10.295 genes codificantes de proteínas, en promedio.
Los científicos compararon sus propios subtipos de CG con los subgrupos de CG en los datos del TCGA, y encontraron que los subgrupos en el TCGA de los subtipos 2 y 4 mostraron las mejores y peores supervivencias, respectivamente. Los CG positivos para MSI y EBV estaban enriquecidos significativamente en el subgrupo similar al subtipo 2, mientras que los CG genéticamente estables se enriquecieron en el subgrupo similar al subtipo 4. Definieron el subtipo 2 como representación de procesos relacionados con la respuesta inmune (presentación de antígenos, señalización del receptor BCR/TNF/tipo Toll, señalización TCR y fagosoma). El subtipo 3 representó de manera única los procesos relacionados con el metabolismo (fosforilación oxidativa, b-oxidación de ácidos grasos y ciclo de citrato). El subtipo 4 representó principalmente los procesos relacionados con la invasión (citoesqueleto de actina y MAPK, PI3K-AKT, WNT, RHOA y señalización de cadherina).
Los autores concluyeron que, en función de sus datos, los subtipos 2 y 4 se pueden caracterizar como tumores inmunogénicos e invasivos con tasas de supervivencia posiblemente buenas y malas, respectivamente, similares a los subgrupos 2 y 4, similares en la cohorte del TCGA. Notaron que los tumores en el subtipo 2 muestran una fuerte actividad inmune que puede contribuir a un buen pronóstico y que los tumores en el subtipo 4 muestran un fuerte potencial de invasión que puede contribuir a un mal pronóstico. El estudio fue publicado el 14 de enero de 2019 en la revista Cancer Cell.
Enlace relacionado:
Universidad de Corea