Cambios en la carga bacteriana y los niveles de citoquinas en suero predicen la probabilidad de morir por sepsis
Por el equipo editorial de LabMedica en español
Actualizado el 01 Jun 2020
Se puede utilizar un método de análisis para la cuantificación sensible y rápida de las citoquinas séricas y la carga bacteriana para predecir la probabilidad de que un paciente muera por sepsis o shock séptico.Actualizado el 01 Jun 2020
La sepsis es causada por una respuesta inmune inflamatoria desencadenada por una infección. Es una afección potencialmente mortal que surge cuando la respuesta del cuerpo a la infección causa daño a sus propios tejidos y órganos. Los signos y síntomas comunes incluyen fiebre, aumento de la frecuencia cardíaca, aumento de la frecuencia respiratoria y confusión. También puede haber síntomas relacionados con una infección específica, como tos en caso de neumonía o dolor al orinar cuando hay una infección renal. En los muy jóvenes, ancianos y aquellos con un sistema inmune debilitado, puede no haber síntomas de una infección específica y la temperatura corporal puede ser baja o normal, en lugar de alta.
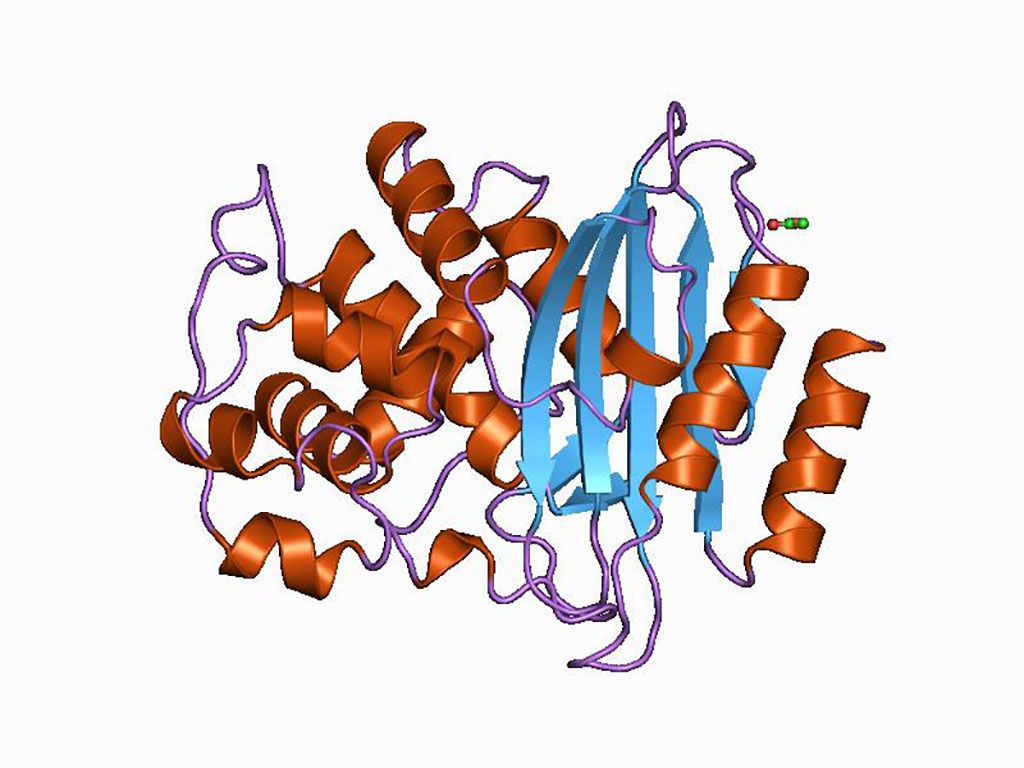
Imagen: Representación en dibujos animados de la estructura molecular de la proteína blaTEM (beta-lactamasa) (Fotografía cortesía de Wikimedia Commons)
Para facilitar el diagnóstico, el seguimiento y el tratamiento de enfermedades infecciosas como las responsables de la sepsis, los investigadores de la Universidad de Chicago (IL, EUA) desarrollaron un método de cuantificación sensible y rápido para la carga bacteriana y las citoquinas séricas a partir de muestras biológicas humanas.
El método utiliza ensayos digitales de ligadura de proximidad (dPLA) para cuantificar marcadores de ácidos nucleicos y proteínas. La lectura de la PCR digital por gotitas (ddPCR), en el protocolo PLA, permitió la medición simultánea de genes 16S rARN específicos de Gram negativos (GN) y Gram positivos (GP) (que reflejan la abundancia de todas las bacterias GN y GP en las muestras de los pacientes) y del gen blaTEM (Beta-lactamasa) (que induce resistencia a los antibióticos Beta-lactámicos) junto con los niveles de proteína IL-6 y TNF-alfa en la misma muestra de los pacientes.
Una ventaja importante de este método de amplificación digital es su capacidad para cuantificar cambios muy pequeños en la concentración de estas moléculas. La ddPCR tiene una resolución de una molécula de ADN único en muestras, y los investigadores pudieron lograr una resolución subfemtomolar para los objetivos de proteínas.
Para demostrar el potencial de este enfoque, los investigadores lo utilizaron primero para analizar muestras de líquido de lavado broncoalveolar (BALF) de pacientes con asma leve a grave, y encontraron que los pacientes con asma tenían niveles más altos de bacterias GN e IL-6 que los individuos de control sanos. Luego utilizaron los ensayos para caracterizar longitudinalmente muestras de plasma de pacientes con shock séptico, revelando varias características moleculares asociadas con la recuperación o la muerte. Los análisis mostraron que los cambios en el tiempo de varios biomarcadores, y no sus concentraciones absolutas, fueron predictores confiables de los resultados de los pacientes. La aplicación del análisis del árbol de decisión a los resultados obtenidos por este método permitió la predicción de la mortalidad del paciente/tasa de recuperación del shock séptico con más del 90% de exactitud.
“Nuestros hallazgos brindan un método nuevo para el diagnóstico de sepsis con el potencial de identificar temprano el patógeno causal”, dijo el autor colaborador, Dr. Gokhan Mutlu, profesor de medicina de la Universidad de Chicago. “Esto nos permitirá usar los antibióticos apropiados antes de que los resultados del cultivo estén disponibles y minimizar el uso de antibióticos necesarios para tratar la infección. Al combinar los datos relacionados con el patógeno y la respuesta del huésped, podemos predecir los resultados en los pacientes con sepsis”.
“Se necesita una prueba rápida como esta en muchas situaciones y realmente podría cambiar el juego para el tratamiento de la sepsis”, dijo el autor principal, el Dr. Savas Tay, profesor asociado de ingeniería molecular en la Universidad de Chicago. “Esta es una enfermedad que puede matar a todos, independientemente de su situación”.
La prueba rápida para la cuantificación digital de citoquinas y bacterias se describió en la edición en línea del 25 de mayo de 2020 de la revista Nature Communications.
Enlace relacionado:
Universidad de Chicago