Cepas de Salmonella resistentes a los medicamentos causan infecciones del torrente sanguíneo
Por el equipo editorial de LabMedica en español
Actualizado el 09 Oct 2019
Salmonella enterica subespecie enterica serovar Typhimurium (conocida como S. typhimurium) y otras cepas de Salmonella no tifoideas son causas comunes de infecciones gastrointestinales en personas que viven en los países industrializados.Actualizado el 09 Oct 2019
Sin embargo, en el África subsahariana (SSA), las infecciones invasivas del torrente sanguíneo por Salmonella no tifoidea (iNTS) son comunes, con un total de alrededor de 3,4 millones de casos anualmente, siendo S. typhimurium responsable de aproximadamente dos tercios de estos casos. La resistencia a los medicamentos ha aumentado en grupos sucesivos de S. typhimurium con el tiempo y la tasa de mortalidad por iNTS puede ser extremadamente alta.
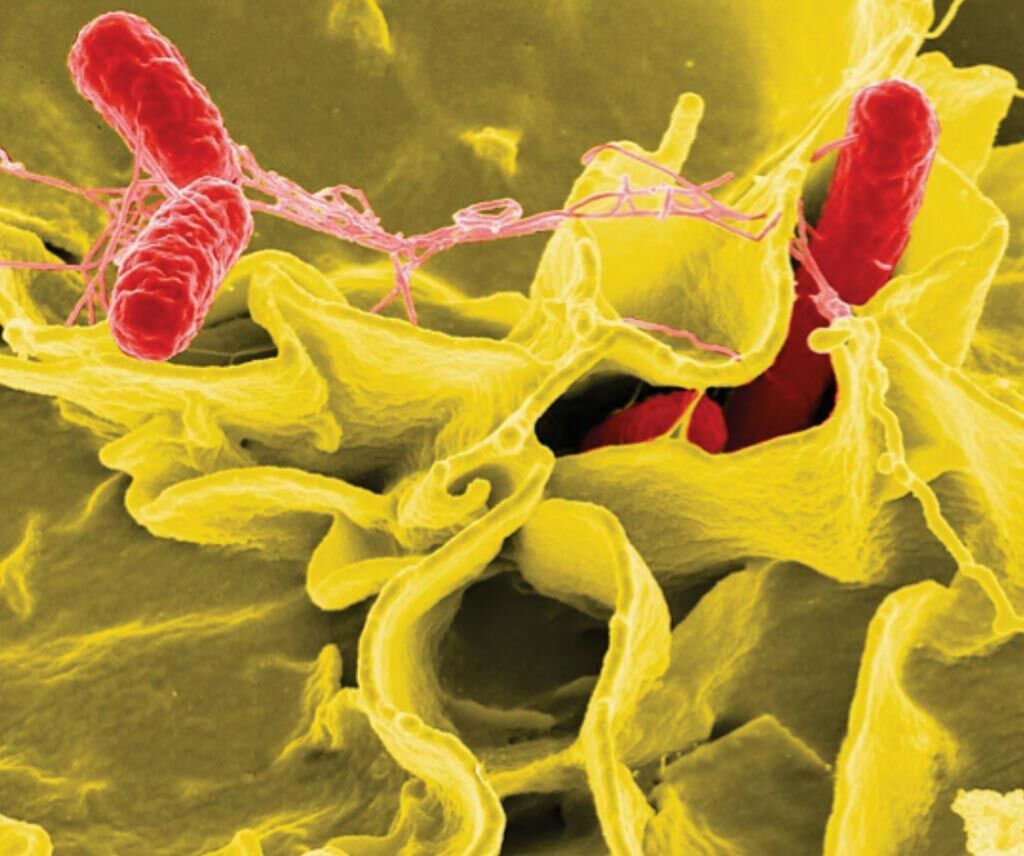
Imagen: Microfotografía electrónica de barrido realzada con color mostrando células humanas cultivadas invadidas por Salmonella typhimurium (rojo) (Fotografía cortesía del Instituto Nacional de Alergias y Enfermedades Infecciosas de los EUA).
Los científicos del Instituto de Medicina Tropical (Amberes, Bélgica) y sus colegas recolectaron muestras de sangre de personas con sospecha de infecciones del torrente sanguíneo en hospitales de la República Democrática del Congo. Se incluyeron todas las cepas de S. typhimurium resistentes a azitromicina (AZI) disponibles para este estudio (n = 54). Se seleccionaron muestras de 27 aislados representativos de S. typhimurium no resistentes a AZI como controles para este análisis.
Los aislamientos, confirmados bioquímicamente como Salmonella spp., fueron serotipifcados utilizando antisueros comerciales (Sifin, Berlín, Alemania). El ADN de las 81 cepas se purificó utilizando el kit Gentra PureGene Yeast/Bact (Qiagen, Hilden, Alemania), siguiendo las pautas del fabricante y el ADN se secuenció en una plataforma Illumina HiSeq (Illumina, San Diego, CA, EUA). El contenido del adaptador Illumina se eliminó de las lecturas usando Trimmomatic v.0.33.
Se sabe que las infecciones por iNTS en el África subsahariana están dominadas por un tipo de S. typhimurium conocido como ST313, asociado con la resistencia a los antibióticos. Dos grupos de ST313 (denominados linaje I y II) se separaron de forma independiente y posteriormente se extendieron por el continente africano. El tiempo ha elevado la resistencia a los antibióticos y el linaje II ahora es la causa principal de las infecciones por iNTS. El análisis de estos genomas de S. typhimurium identificó un nuevo subgrupo que se ramifica de ST313, llamado linaje II.1. Se estima que este subgrupo, que surgió en 2004, muestra una extensa resistencia a los medicamentos (XDR).
Sandra Van Puyvelde, PhD, profesora asistente y primera autora del estudio, dijo: “Todos los genes de resistencia a los antibióticos que contribuyen a ‘XDR’ están presentes en el mismo plásmido. Esto es preocupante porque un plásmido es un elemento genético móvil que se podría transferir a otras bacterias. Mientras acumulamos más resistencia a los antibióticos, descubrimos que la nueva línea de Salmonella typhimurium también muestra más cambios genéticos y de comportamiento que sugieren una evolución continua de las bacterias hacia las infecciones del torrente sanguíneo”. El estudio fue publicado el 19 de septiembre de 2019 en la revista Nature Communications.
Enlace relacionado:
Instituto de Medicina Tropical
Sifin
Qiagen
Illumina