Comparan métodos de secuenciación para la genotipificación del virus de la hepatitis C
Por el equipo editorial de LabMedica en español
Actualizado el 09 Sep 2019
Debido a su alta tasa de mutación, el virus de la hepatitis C (VHC) forma cuasiespecies virales, clasificadas de acuerdo con las regiones altamente variables de la proteína de la envoltura y de la proteína 5B no estructural (NS5B). Como resultado de esta gran variabilidad genética, se han identificado siete genotipos y más de 67 subtipos.Actualizado el 09 Sep 2019
La genotipificación del VHC es parte de la evaluación de pacientes recién diagnosticados y siempre ha sido importante para guiar el tratamiento. Teniendo en cuenta que el genotipo es un elemento clave en el tratamiento de pacientes infectados con el VHC, se recomienda la determinación del genotipo. La técnica utilizada con más frecuencia para genotipificar el VHC es la reacción en cadena de la polimerasa cuantitativa en tiempo real (qRT-PCR). Esta técnica no puede proporcionar un genotipo/subtipo preciso para muchas muestras.
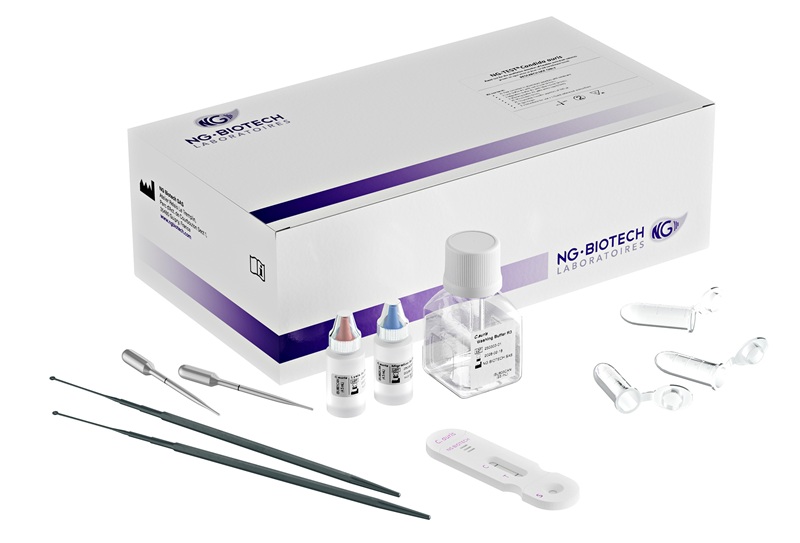
Imagen: El ensayo de sonda VERSANT HCV Genotype 2.0 Line (Fotografía cortesía de Siemens Healthcare Diagnostics).
Los microbiólogos y sus colegas que trabajan en la Universidad Católica de Lovaina (Bruselas, Bélgica) desarrollaron un método interno con el objetivo de identificar con exactitud el genotipo de todas las muestras. El equipo analizó 100 muestras de pacientes infectados con VHC mediante el ensayo VERSANT HCV Genotype 2.0 LiPA (Siemens Healthcare Diagnostics, Erlangen, Alemania), un ensayo que cubre los tipos y subtipos frecuentes de VHC. Posteriormente, se realizó la secuenciación casera NS5B, 5'UTR y Core en estas muestras. Las secuencias obtenidas se compararon con el banco BLAST genómico de VHC.
Los investigadores informaron que las muestras pudieron ser caracterizadas por el ensayo VERSANT LiPA (ocho G1a, 17 G1b, seis G2, 11 G3, 13 G4 y 10 G6). No fue posible discriminar entre G6 y G1 por el ensayo VERSANT LiPA para ocho muestras y 27 tenían un genotipo indeterminado. Cuarenta y una muestras fueron secuenciadas para las tres regiones: NS5B, 5'UTR y Core. Veintitrés muestras fueron secuenciadas para dos regiones: 5 'UTR y Core y 36 muestras fueron secuenciadas solo para NS5B. De las 100 muestras incluidas, 64 muestras fueron analizadas para la secuenciación 5'UTR y Core y 79 muestras fueron analizadas para la secuenciación NS5B. La concordancia general entre el ensayo VERSANT LiPA y la secuenciación, fue superior al 95%.
Los autores concluyeron que la alta variabilidad genética del VHC hace que sea un desafío determinar correctamente el genotipo y los subtipos mediante ensayos comerciales. Para las muestras indeterminadas, se requieren pruebas complementarias. Describieron métodos nuevos y originales de secuenciación de las regiones 5'UTR y Core para confirmar los genotipos de VHC no discriminados por un ensayo comercial, en particular mediante el uso de amplicones ya obtenidos por el Ensayo de sonda de línea VERSANT HCV Genotype 2.0. El estudio fue publicado el 22 de agosto de 2019 en la revista BMC Infectious Diseases.
Enlace relacionado:
Universidad Católica de Lovaina
Siemens Healthcare Diagnostics