Diagnóstico SHERLOCK se optimiza para detección viral rápida
Por el equipo editorial de LabMedica en español
Actualizado el 29 May 2018
Una nueva técnica permite a SHERLOCK detectar un virus directamente en los fluidos corporales, eliminando un paso que requería equipo de laboratorio y expandiendo el potencial de la plataforma para rastrear agentes patógenos como el Zika durante un brote, de manera rápida y barata.Actualizado el 29 May 2018
La plataforma ahora se puede usar para detectar virus directamente en muestras clínicas, como sangre o saliva, eliminando un paso de procesamiento que anteriormente requería un entorno de laboratorio y personal capacitado profesionalmente. El desarrollo prepara a SHERLOCK para uso en áreas donde el entrenamiento especial y los laboratorios clínicos pueden ser un desafío para el acceso.
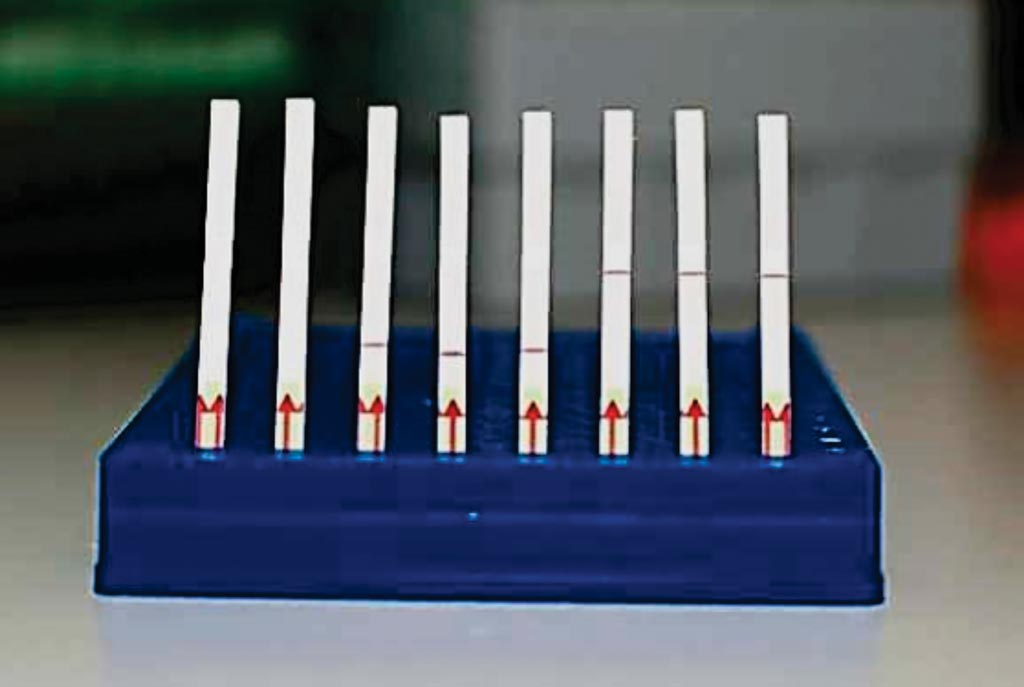
Imagen: Una colección de tiras de papel de análisis, SHERLOCK: tiras de papel no utilizadas (izquierda); tiras de papel que muestran una lectura de SHERLOCK negativa (Mitad); Tiras en papel que muestran una lectura SHERLOCK positiva (derecha) (Fotografía cortesía del Instituto Broad de MIT y Harvard).
Científicos del Instituto Broad del Instituto Tecnológico de Massachusetts (MIT) y la facultad de medicina de Harvard (Cambridge, MA, EUA) también optimizaron las capacidades de SHERLOCK (sigla en inglés para Specific High-sensitivity Enzymatic Reporter unLOCKing [Alta Sensibilidad Específica Reportero Enzimático desbloqueo)] para diferenciar especies virales relacionadas entre sí y demostró la capacidad de la plataforma para identificar mutaciones clínicamente relevantes, como una pequeña mutación en el virus del Zika que se ha asociado con la microcefalia.
La plataforma de diagnóstico SHERLOCK utiliza una enzima Cas13 programada emparejada con moléculas reporteras para indicar la presencia de un objetivo genético, como un virus. Hasta ahora, un paso preliminar crucial para SHERLOCK consistía en extraer y aislar ácidos nucleicos de muestras de pacientes, lo que generalmente requiere un laboratorio y personal capacitado, por lo que su realización en el campo es difícil.
El equipo desarrolló un método más simple que permite a Cas13 detectar su objetivo directamente en muestras de fluidos corporales como la saliva o la sangre. El proceso se llama HUDSON, [por sus siglas en inglés para calentamiento de muestras de diagnóstico no expuestas para anular las nucleasas (Heating Unextracted Diagnostic Samples to Obliterate Nucleases)]. Consiste en un tratamiento químico y térmico rápido utilizado en las muestras para inactivar ciertas enzimas que, de otra manera, degradarían los objetivos genéticos. Las muestras clínicas procesadas se pueden procesar a través del procedimiento SHERLOCK y los resultados finales de detección, positivos o negativos, se pueden ver fácilmente en la tira de papel. El procedimiento completo se puede completar en menos de dos horas.
A través de la combinación de HUDSON y SHERLOCK, el equipo pudo detectar el virus del dengue directamente en muestras de saliva y suero sanguíneo de pacientes. La plataforma también podría detectar partículas del virus del Zika que se habían agregado a muestras sanas de sangre y orina. Además, el equipo diseñó los reactivos SHERLOCK que hacen que sea aún más fácil y rápido diferenciar varias especies virales relacionadas (Zika, Dengue, Nilo Occidental y fiebre amarilla) entre sí. Estas mejoras son particularmente útiles cuando un paciente tiene síntomas generales, como fiebre, que podrían ser causados por más de un virus.
Pardis C. Sabeti, DPhil, MD, profesor y autor principal del estudio, dijo: “Las herramientas rápidas y sensibles son fundamentales para diagnosticar, vigilar y caracterizar una infección. Hemos tomado la tecnología SHERLOCK y la hemos optimizado en el contexto de estos escenarios biológicos reales aplicados”. El estudio fue publicado el 27 de abril de 2018 en la revista Science.










 (3) (1).png)