Asocian mutaciones de Mycobacterium con resistencia a la claritromicina
Por el equipo editorial de LabMedica en español
Actualizado el 29 Jan 2018
La bacteria Mycobacterium abscessus es la principal causa de enfermedades respiratorias y de infecciones de los tejidos blandos y tiene una resistencia natural a muchos antibióticos in vitro, lo que complica el tratamiento clínico y conduce a resultados insatisfactorios.Actualizado el 29 Jan 2018
Los macrólidos, como la claritromicina y la azitromicina, son considerados como la base del régimen terapéutico para las infecciones complejas por M. abscessus. Sin embargo, la resistencia a la claritromicina está aumentando dramáticamente entre el complejo de M. abscessus.
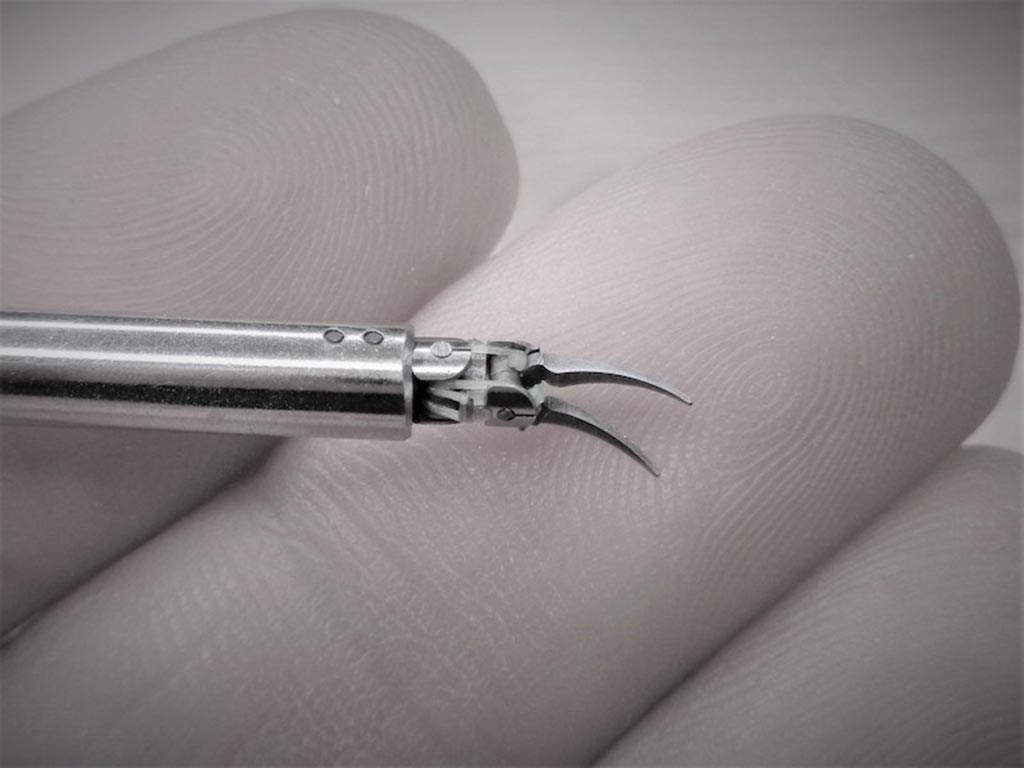
Imagen: El sistema de lectura de placa fluorométrica automatizado, Sensititre OptiRead, para uso con placas de microtitulación Sensititre en las pruebas de susceptibilidad a los antibióticos (Fotografía cortesía de Thermo Fisher Scientific).
Los microbiólogos de la Facultad de Medicina de la Universidad de Tongji (Shanghái, China), recolectaron muestras del Hospital Pulmonar afiliado de Shanghái y se usaron una colección de 139 aislados clínicos de M. abscessus subsp. abscessus y 36 de M. abscessus subsp. massiliense para explorar la utilidad de la técnica de la reacción en cadena de la polimerasa-electroforesis en gel de gradiente de desnaturalización (PCR-DGGE) para la identificación rápida del ARNr 23S (Adenina (2058) -N (6)) - metiltransferasa Erm (41) (erm (41) y las mutaciones del gen 23S rARN (rrl).
El equipo diseñó cuatro conjuntos de cebadores para buscar mutaciones en los genes erm (41) y rrl usando DGGE. Las reacciones de PCR y la electroforesis, así como la recuperación de las bandas y el análisis en gel, se realizaron según los protocolos descritos previamente. Se usó un panel Sensititre RAPMYCO (TREK Diagnostic Systems, East Grinstead, Reino Unido) para probar la susceptibilidad de varios antibióticos, incluida la claritromicina contra los aislados del complejo 175 M. abscessus.
Los autores encontraron una combinación de 16 patrones diferentes de DGGE para el gen erm (41), incluyendo 16 en M. abscessus subsp. abscessus y uno en M. abscessus subsp. massiliense. Se obtuvieron seis patrones de DGGE para el gen rrl. Las mutaciones en los genes erm (41) y rrl detectadas por DGGE fueron 100% idénticas a las mutaciones detectadas por secuenciación de ADN. Entre los 139 aislados de M. abscessus subsp. abscessus, 36 aislamientos exhibieron resistencia a la claritromicina en el día 5. Los otros 103 fueron sensibles a la claritromicina en el día 5 y 83 de estos mostraron resistencia inducible después de 14 días de incubación. De los 36 aislados de M. abscessus subsp. massiliense, 27 fueron sensibles y ocho fueron resistentes a la claritromicina en los días 5 y 14. Cabe destacar que uno de los aislados fue sensible a la claritromicina el día 5 pero mostró resistencia inducible el día 14.
Los autores concluyeron que sus resultados demostraron que la tecnología DGGE es muy sensible a las mutaciones puntuales, así como a las pequeñas inserciones y deleciones en las secuencias de ADN. La exactitud fue esencialmente del 100% entre los fragmentos dirigidos de los genes erm (41) y rrl. Los resultados de la secuenciación del genoma completo confirmaron la eficacia de DGGE; se detectaron todos los polimorfismos de nucleótidos distintos. La PCR basada en DGGE es una técnica práctica para la detección rápida de mutaciones en los genes erm (41) y rrl, asociados con la resistencia a la claritromicina en el complejo de M. abscessus. El estudio fue publicado en la edición de diciembre de 2017 de la revista Journal of Microbiological Methods.