Evalúan prueba molecular para H. pylori
Por el equipo editorial de LabMedica en español
Actualizado el 26 Jan 2018
Actualizado el 26 Jan 2018
El Helicobacter pylori es una bacteria Gram-negativa que puede causar infecciones estomacales crónicas, que se conoce como la causa principal de úlceras gástricas y, en consecuencia, para el desarrollo de cáncer gástrico si no se trata.
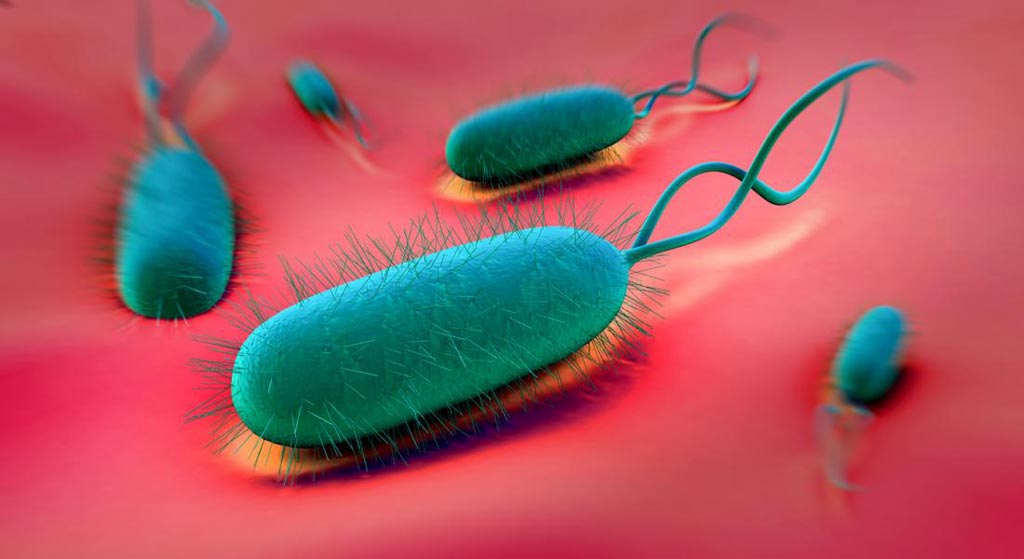
Imagen: Una interpretación artística de H. pylori (Fotografía cortesía de Medical News Today).
En tiempos de aumento de la resistencia a los antibióticos, la necesidad de procedimientos de cribado de resistencia más rápidos que puedan reemplazar los métodos de cultivo convencionales gana importancia ya que H. pylori es una bacteria fastidiosa y de crecimiento lento que puede hacer que las pruebas de susceptibilidad con cultivos sean una tarea lenta y desafiante.
Los microbiólogos médicos de la Universidad de Zúrich (Suiza; www.uzh.ch) utilizaron un conjunto de 60 cepas de H. pylori de la colección de cultivos de su Instituto de Microbiología Médica. Estas cepas se aislaron entre 2013 y 2015 a partir de biopsias gástricas que habían sido enviadas al laboratorio para la realización de pruebas de susceptibilidad después del fracaso del tratamiento con claritromicina (CLR). Las cepas se seleccionaron intencionadamente en función de su estado de resistencia fenotípica para obtener un número uniformemente distribuido de cepas susceptibles a la CLR (concentraciones mínimas inhibitorias (MIC) ≤0,25 mg/L) y de cepas resistentes a la CLR (CIM de 0,016 a 256 mg/L).
Los científicos compararon las pruebas de susceptibilidad de la CLR con E-Test (bioMérieux, Marcy l'Etoile, Francia) con el ensayo de reacción en cadena de la polimerasa Lightmix en tiempo real (RT-PCR, Roche, Basilea, Suiza). Se usó un termociclador Veriti (Applied Biosystems, Forster City, CA, EUA) para el programa de secuenciación de ciclos. El equipo encontró una alta concordancia (95%) entre la detección fenotípica de resistencia a la CLR mediante E-Test y la detección realizada con el Lightmix RT-PCR. Los resultados discrepantes se verificaron mediante la secuenciación del gen 23S rARN que siempre confirmó los resultados obtenidos usando la prueba Lightmix RT-PCR. Más aún, se pudo detectar H. pylori en nuestras de biopsias y de heces usando la Lightmix RT-PCR que pudo identificar el genotipo correcto de H. pylori.
Los autores concluyeron que sus datos demostraron que existe una asociación fuerte entre las mutaciones específicas en el 23S rADN y la resistencia a la CLR. La RT-PCR demostró ser una herramienta excepcional para una detección rápida y confiable de H. pylori. Además, permite la determinación de la resistencia a la CLR unas pocas horas antes de la formulación de un tratamiento con antibióticos y debe reducir drásticamente los casos de fracaso de estos. El estudio fue publicado en la edición de enero de 2018 de la revista Diagnostic Microbiology and Infectious Disease.






 Analyzer.jpg)