Marcador viral predice gravedad de gripe en pacientes infectados
Por el equipo editorial de LabMedica en español
Actualizado el 12 Dec 2017
Actualizado el 12 Dec 2017
Los virus de la influenza A son los agentes causantes de epidemias anuales, brotes zoonóticos esporádicos y ocasionalmente pandemias. En todo el mundo, las infecciones respiratorias agudas causadas por el virus de influenza A siguen siendo una de las principales causas de enfermedad aguda y muerte.

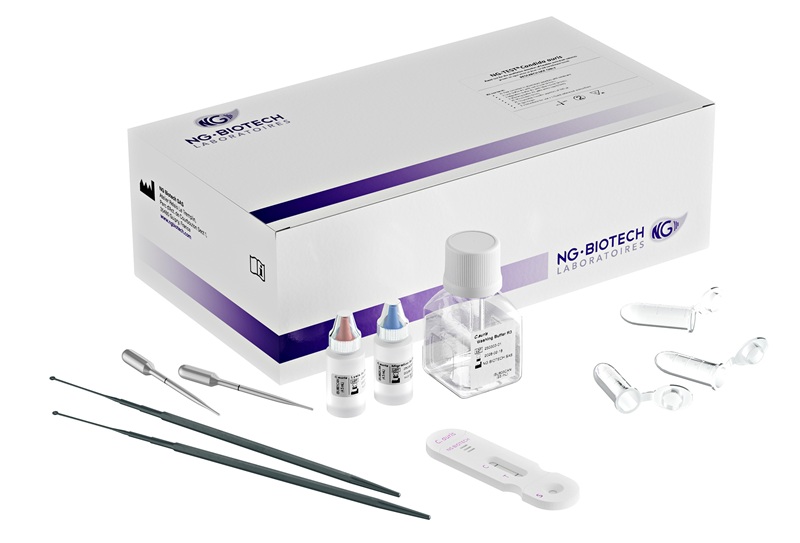
Imagen: El Bioanalizador Agilent 2100 es una plataforma basada en microfluidos para el dimensionamiento, cuantificación y control de calidad de ADN, ARN, proteínas y células (Fotografía cortesía de CORELAB).
La influenza es particularmente peligrosa para los bebés, los ancianos y las personas con problemas médicos subyacentes, pero las personas sanas a veces también experimentan una infección grave. Se han encontrado marcadores de gravedad para cepas específicas, pero un marcador general que se pudiera aplicar a cepas múltiples sería más útil para informar el tratamiento y la política.
Un equipo de científicos del Centro Nacional de Biotecnología de España (Madrid, España) incluyó en su estudio a pacientes de una cohorte grave/fatal que tenían casos confirmados de influenza A (H1N1)pdm09, tenían más de cuatro años y eran menores de 65 años, que habían ingresado en la unidad de cuidados intensivos (UCI) y con la información relacionada con los factores de riesgo que se reflejaban en su historia clínica. Aquellos pacientes que desarrollaron una enfermedad muy grave no mostraron ninguna comorbilidad asociada con la infección grave por el virus de la influenza A, y los pacientes fallecidos presentaron algunas enfermedades comórbidas. Los pacientes leves fueron casos confirmados de influenza A (H1N1)pdm09, mayores de cuatro años y menores de 65 años, que desarrollaron una enfermedad leve y fueron controlados por los centros médicos centinela.
El equipo se centró en genomas virales defectuosos (DVG), que consisten en fragmentos de ARN viral con información genética faltante, que se encuentran en múltiples cepas del virus de la influenza. Para probar si los DVG pueden servir como un marcador general de la gravedad de la influenza, infectaron cultivos de células tisulares humanas y de ratones con diferentes cepas del virus de la gripe A H1N1, el subtipo responsable de las pandemias de influenza.
El equipo también analizó los genomas de virus aislados de muestras respiratorias tomadas de personas que experimentaron una infección grave o muerte durante la pandemia de “gripe porcina” de 2009 o las estaciones posteriores de “gripe porcina”. La identidad de los virus mutantes rescatados se determinó por secuenciación del ADN derivado de los genes de la polimerasa segmentos de ARN PA y PB2 por amplificación por PCR de transcripción inversa (RT-PCR). La calidad y la cantidad de cada preparación de ARN se controlaron usando el instrumento Agilent 2100 Bioanalyzer (Agilent Technologies, Santa Clara, CA, EUA). El análisis de citometría de flujo se realizó en un citómetro LSR II (BD Biosciences, San José, CA, EUA).
El equipo descubrió que la cepa H1N1 que causaba síntomas graves tenía una acumulación de DVG significativamente menor que las cepas de influenza A de la que se tomaron muestras de personas que solo experimentaron síntomas leves. En conjunto, estos resultados sugieren que los niveles bajos de DVG pueden indicar un mayor riesgo de enfermedad grave en pacientes infectados con el virus de la gripe A. Con estudios adicionales, estos hallazgos podrían ayudar a predecir la gravedad de la influenza, guiar el tratamiento del paciente e informar las estrategias de prevención de la influenza. El estudio fue publicado el 12 de octubre de 2017 en la revista Public Library of Sciences Pathogens.