Secuenciación de ADN del SARM predice la supervivencia de los pacientes
Por el equipo editorial de LabMedica en español
Actualizado el 31 Aug 2017
Actualizado el 31 Aug 2017
El Staphylococcus aureus resistente a la meticilina (SARM) es una bacteria que se ha convertido en resistente a la mayoría de los tipos de antibióticos, y mueren hasta el 20% de los pacientes que padecen infecciones invasivas. Aunque S. aureus es una bacteria común que vive en la piel, si entra dentro del cuerpo a través de un corte puede causar septicemia.
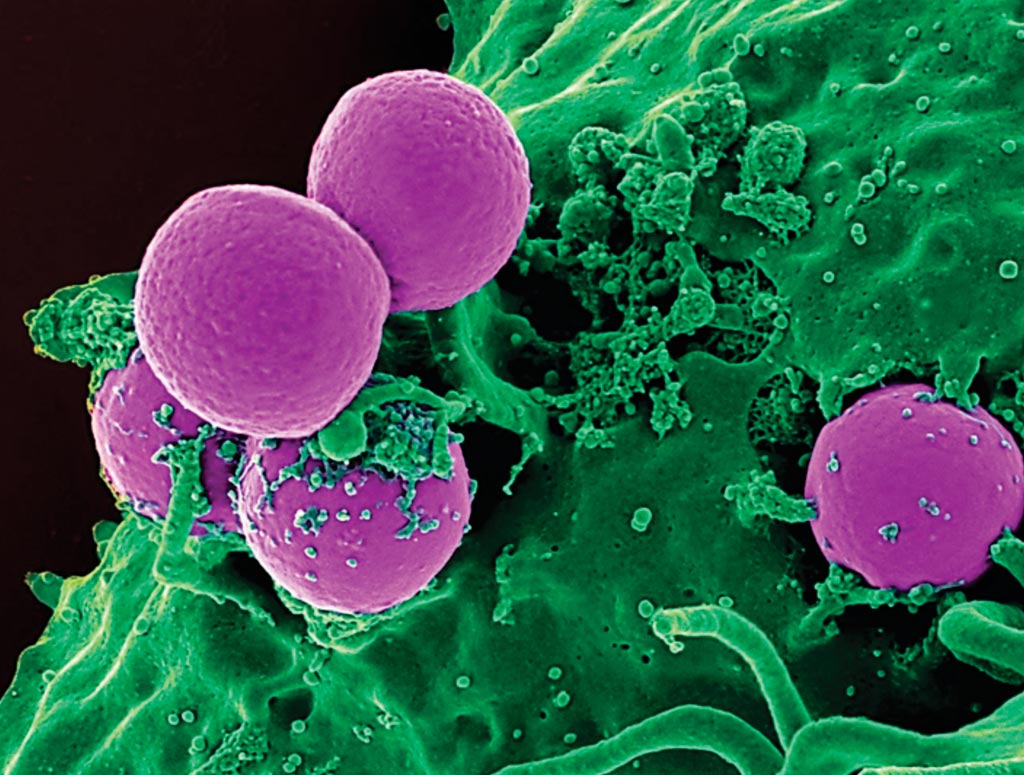
Imagen: Una microfotografía electrónica de barrido (SEM) de un neutrófilo humano ingiriendo un Staphylococcus aureus resistente a la meticilina (SARM) (Fotografía cortesía del Instituto Nacional de Alergia y Enfermedades Infecciosas).
Secuenciar el ADN de la superbacteria SARM puede identificar con exactitud a los pacientes con mayor riesgo de muerte y podría ayudar a los profesionales médicos a desarrollar nuevos tratamientos a medida que avanzan hacia la medicina personalizada. Esta infección, potencialmente mortal, puede afectar a miles de pacientes cada año en el Reino Unido. Hay dos cepas principales de SARM, que se encuentran en los hospitales del Reino Unido, llamadas CC22 y CC30.
Un equipo de científicos liderados por los del Centro Milner para la Evolución, de la Universidad de Bath (Reino Unido), pudieron estudiar muestras de sangre de alrededor de 300 pacientes con septicemia, observando cómo se comportaron las diferentes cepas de SARM y evaluando su letalidad. La secuenciación del ADN se realizó junto con la medición de la toxicidad, o la capacidad de matar células humanas, de las cepas de SARM, así como su capacidad para formar biopelículas peligrosas. Las biopelículas se forman cuando grupos de bacterias secretan proteínas que las pegan y recubren las superficies con una sustancia resbaladiza. Las biopelículas hace que sea más fácil, para las bacterias, evadir el sistema inmunológico del paciente y, además, puede bloquear la acción de los antibióticos. Son un problema, especialmente, en los pacientes que utilizan catéteres caso en que hasta la mitad de los pacientes pueden contraer una infección.
Los científicos examinaron el código genético de la bacteria infectante SARM y compararon esta información con los factores de riesgo individuales para cada paciente, incluyendo la edad, la presencia de cualquier otra enfermedad y señalaron si el paciente seguía vivo después de 30 días de la infección y, en caso de haber fallecido, si SARM había contribuido a su muerte. Encontraron que, para las cepas CC22, tanto su toxicidad como su capacidad de formación de biopelículas desempeñaban un papel importante en si el paciente sobrevivió a su infección. Sin embargo, estos no parecen estar involucrados en el resultado de los pacientes que habían sido infectados con las cepas CC30, lo que significa que esta cepa está matando a las personas de una manera diferente.
Ruth C. Massey, PhD, una conferencista experimentada, quien lideró el estudio, dijo: “Desafortunadamente el 20% de los pacientes con septicemia mueren y los casos están en aumento, lo que sugiere que el control de la infección y las opciones de tratamiento existentes, son insuficientes para hacer frente a este importante problema de salud”. “Hemos identificado que SARM mata a las personas de diferentes maneras dependiendo de la cepa, y que las cepas de baja toxicidad, CC30, matan a los pacientes usando un mecanismo aún desconocido. Podría ser que sean mejores en evadir el sistema inmune”. El estudio se publicó el 7 de agosto de 2017, en la revista Nature Microbiology.