Software para análisis de imágenes identifica muestras positivas para SARM
Por el equipo editorial de LabMedica en español
Actualizado el 30 Mar 2016
Actualizado el 30 Mar 2016
En un estudio de microbiología digital de casi 60.000 muestras, un software nuevo, WASPLab, desarrollado para realizar la puntuación automatizada de colonias provenientes de los cultivos en medios cromogénicos, proporcionó una detección superior del Staphylococcus aureus resistente a la meticilina (SARM).
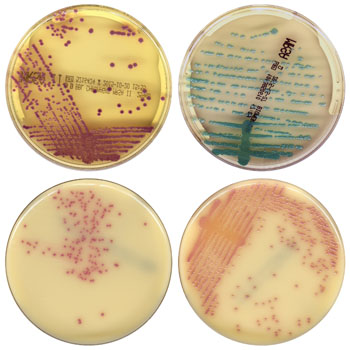
Imagen: Una puntuación automatizada de las colonias cultivadas en placas con medios cromogénicos para la detección del SARM utilizando el software de análisis de imágenes, WASPLab (Fotografía cortesía del Grupo COPAN/PRNewsFoto).
Los revolucionarios resultados, proceden de un estudio multicéntrico internacional, que analizó 57,690 muestras, en las que el software de la plataforma WASPLab “Módulo de Detección Cromogénica” (MDC) de COPAN Diagnostics, Inc. (Murrieta, CA, EUA), detectó y separó las muestras positivos de las muestras negativas de SARM examinando los agares cromogénicos, con una sensibilidad del 100% y una especificidad del 90% -96% (que variaba según la ubicación). El software detectó 153 pacientes adicionales con SARM positivos que se habían pasado por alto cuando se usó la lectura manual.
El tamaño de la muestra del estudio y el nivel de sensibilidad del 100%, “hace que este estudio no tenga precedentes en la industria”, dijo Norman Sharples, director ejecutivo de Copan Diagnostics.
El MDC utiliza un algoritmo para identificar la presencia de colonias en un cultivo y diferenciar entre los varios colores que se agrupan en los cultivos negativos y positivos. El estudio se centró en la identificación de SARM utilizando agar cromogénico entre 3 diferentes fabricantes (BD Diagnostics, bioMérieux y Bio-Rad Laboratories) que produjeron un crecimiento con diferentes colores de pigmentación. Los cultivos fueron leídos, automáticamente por WASPLab MDC y, de forma manual, por un profesional de laboratorio. Los profesionales de laboratorio no tenían conocimiento de los resultados del software.
El umbral de lectura del MDC fue establecido intencionadamente con un alto nivel de prudencia diseñado para que no se pasara por alto, ningún cultivo positivo para el SARM. De hecho, en este amplio estudio, el MDC nunca reportó cultivos positivos manuales, como negativo, lo que demuestra una sensibilidad del 100%. La puntuación de 90% -96% de especificidad varió, según el lugar, debido al umbral conservador de la configuración del MDC, que establece las limitaciones bajas con el fin de evitar cualquier interpretación de “falso negativo”. Lo más importante, los datos de este estudio, sugieren que el software MDC es un poco más sensible que la lectura manual de las placas de cultivo.
La capacidad del software reduce el tiempo de operario, considerablemente”, dijo el Dr. Sharples, “El software no está destinado a ser utilizado sin el tecnólogo, sino para proporcionarle al técnico con herramientas valiosas que re-analicen rápidamente los cultivos, detecten la presencia de organismos de interés concretos o patógenos conocidos, y luego clasifique los cultivos positivos y negativos en grupos, lo que permite tiempos de respuesta más rápidos y resultados para poder tomar acciones concretas dentro de la ventana terapéutica”.
Los resultados de los datos muestran que el WASPLab CDM se puede utilizar para clasificar con exactitud, independientemente del sustrato cromogénico. Su aplicación para el SARM es sólo el primer paso, en una serie de algoritmos desarrollados por COPAN y que están a disposición para los usuarios WASPLab con el fin de promover la pertinencia y la velocidad de los resultados de laboratorio.
“La microbiología clínica se basa, en gran medida, en las habilidades de lectura y de interpretación del técnico y del microbiólogo. En un momento de disminución de los recursos y el aumento de los volúmenes de trabajo, el avance de estos algoritmos de imagenología, apoyan el valor de la microbiología digital y de la automatización total del laboratorio para la salida continua de resultados de calidad y la mejora de la atención a los pacientes”, concluyó el Dr. Sharples.
El estudio, realizado por Faron ML et al., fue publicado en línea, adelante de impresión, el 30 de diciembre de 2015, en la revista Journal of Clinical Microbiology.
Enlace relacionado:







 Analyzer.jpg)