Novedoso análisis molecular permite diagnosticar ocho parásitos gastrointestinales
Por el equipo editorial de LabMedica en español
Actualizado el 12 Sep 2013
Se ha desarrollado una plataforma rápida con reacción en cadena de polimerasa cuantitativa (qPCR) multiplex en paralelo, en tiempo real y de alto rendimiento, para el diagnóstico de parásitos gastrointestinales.Actualizado el 12 Sep 2013
El diagnóstico de los parásitos gastrointestinales se ha basado tradicionalmente en la microscopía de heces, la cual tiene bajas sensibilidad y especificidad diagnósticas puesto que la capacidad del microscopista para detectar los parásitos está directamente relacionada con el número de organismos en las heces.
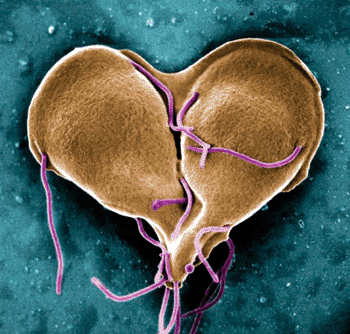
Imagen: Una microfotografía electrónica de barrido (SEM) del parásito intestinal, Giardia lamblia (Fotografía cortesía del Dr. Stan Erlandsen).
Los helmintólogos de los Institutos Nacionales de Salud de los EUA (Bethesda, MD, EUA) trabajando con otros, recogieron muestras de heces de 400 niños asintomáticos, de 13 meses de edad, que viven en Ecuador. Los niños formaban parte de un estudio de cohorte más amplio en curso, en el cual la microscopía directa y el método de Kato-Katz fueron realizados en el momento de recolección de las heces. A un segundo grupo de 125 niños asintomáticos de ocho a 14 años de edad también se les recogieron muestras fecales como parte de un estudio separado para un tratamiento antihelmíntico.
Se utilizaron cebadores y sondas específicas para cada especie, con el fin de detectar ocho parásitos patogénicos gastrointestinales comunes: Ascaris lumbricoides, Necator americanus, Ancylostoma duodenale, Giardia lamblia, Cryptosporidium spp, Entamoeba histolytica, Trichuris trichiura y Strongyloides stercoralis. Todas las qPCR fueron realizadas en placas ópticas de 96 pozos, MicroAmp (Applied Biosystems, Foster City, CA, EUA) y las muestras fueron procesadas en el sistema ABI 7900HT Fast Real-Time PCR de Applied Biosystems y se compararon con una lámina estándar de montaje húmedo directo para microscopía de heces.
La qPCR mostró mayores tasas de detección para todos los parásitos en comparación con la microscopía directa. Por ejemplo, el análisis molecular identificó Ascaris en 28 muestras positivas de 400 (7,0%) mientras que los frotis directos identificaron 22 muestras positivas de 400 (5,5%). La diferencia entre la qPCR y la microscopía se observó incluso más dramáticamente para G. lamblia con 103 muestras positivas adicionales para este parásito, y 126 de 400 (31,5%) con qPCR en comparación con 23 de 400 (5,8%) por microscopía. La qPCR permitió diferenciar entre los pacientes sin parásitos y los que tenían poliparasitismo con mayor exactitud y con mayores tasas de detección que la microscopía directa.
Los autores llegaron a la conclusión de que el sistema de alto rendimiento que se estaba probando en el campo, en una zona de recursos limitados, proporciona un método sensible y específico para detectar el parasitismo y el poliparasitismo gastrointestinal, con amplias implicaciones para la comunidad en cuanto al tratamiento y a los métodos para evaluar la eficacia del tratamiento. El estudio fue publicado en la edición de agosto de 2013 de la revista American Journal of Tropical Medicine and Hygiene.
Enlaces relacionados:
US National Institutes of Health
Applied Biosystems








 (3) (1).png)