Método más rápido identifica cepas de Salmonella
Por el equipo editorial de LabMedica en español
Actualizado el 22 Jul 2013
Un método de subtipificación basado en secuencia, puede reducir en más de la mitad el tiempo necesario para que los funcionarios de salud identifiquen las cepas de Salmonella.Actualizado el 22 Jul 2013
Se requieren métodos rápidos, eficientes y exactos para la identificación de cepas específicas de la serovar Newport, durante el brote, permitiendo a los investigadores epidemiológicos identificar las cepas exactas de Salmonella que hacen que las personas se enfermen, para encontrar y eliminar más rápidamente el origen de la enfermedad.
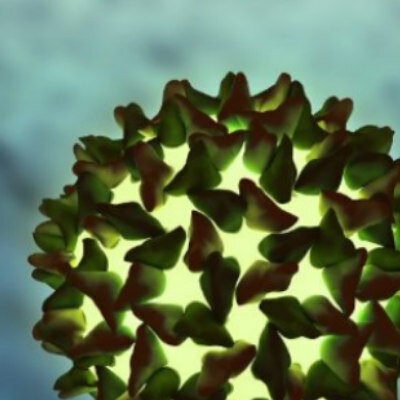
Imagen: Microfotografía electrónica de barrido (SEM) de la bacteria Salmonella (Fotografía cortesía de los Centros para el Control de Enfermedades de los EE.UU).
Los microbiólogos de la Universidad Estatal de Pennsylvania (White Oak, PA, EUA) desarrollaron un nuevo método para identificar las cepas de Salmonella enterica subespecie enterica serovar Newport. El método se centra en dos genes de virulencia y dos regiones nuevas de ADN de la Salmonella llamadas repeticiones palindrómicas, cortas, interespaciadas, agrupadas regularmente, o CRISPRs. Los científicos idearon un método de tipificación de secuencia multi-virulencia-locus o MVLST, que puede detectar las diferencias específicas de las cepas en el ADN de estas cuatro ubicaciones. El método se denomina CRISPR-MVLST.
Se obtuvieron un total de 84 aislamientos clínicos de la serovar Newport y se seleccionaron sistemáticamente a lo largo de cinco años. Los aislamientos que se recibieron más cercanos al día 1 y el día 15 de cada mes, desde 2007 hasta 2011 fueron seleccionados para representar una colección imparcial de los aislados clínicos humanos. Los investigadores usaron amplificación de reacción en cadena de la polimerasa (PCR), secuenciación del ADN y análisis de la secuencia y el tipo de asignación de secuencias, para estudiar los aislamientos. Hacia el final del proyecto, aplicaron su análisis a un brote de Salmonella, asociado con tomates que sucedió en Pennsylvania, en el verano de 2012, en el que 37 personas se enfermaron.
Los autores llegaron a la conclusión de que las ventajas de CRISPR-MVLST son la automatización potencial del procedimiento, la capacidad para llevar a cabo este análisis en una forma de alto rendimiento, y que los datos de secuencia son inherentemente más manejable para aplicaciones posteriores tales como análisis filogenéticos y evolutivos en comparación con las electroforesis en gel de campo pulsado (PFGE).
Nikki Shariat, PhD, autora principal del estudio, dijo: “En este momento, los laboratorios de salud pública utilizan una técnica llamada PFGE, para hacer la sub-tipificación de las cepas de Salmonella, y la identificación se tarda, normalmente, entre uno y tres días para una cepa específica. La técnica que ideamos, a menudo, toma un solo día. El cincuenta por ciento de las bacterias tiene regiones CRISPR y el uso de éstas para la identificación se ha hecho con un buen número de bacterias, tales como Mycobacterium tuberculosis, así como con algunos que causan enfermedades transmitidas por alimentos, tales como Campylobacter y Escherichia coli”. El estudio fue publicado el 15 de mayo de 2013, en la revista Journal of Clinical Microbiology.
Enlace relacionado:
The Pennsylvania State University