Nuevo modelo computacional mejora significativamente calidad de imágenes de microscopía
Actualizado el 26 Dec 2024
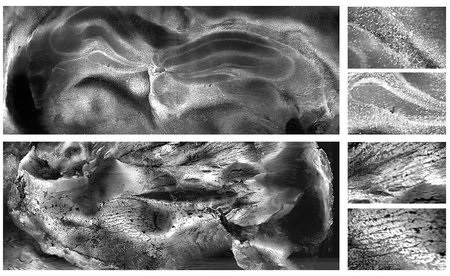
El procesamiento computacional de imágenes permite el examen detallado de muestras utilizando diversos microscopios ópticos. Si bien se han logrado avances significativos en este campo, aún existe potencial para mejorar aspectos como el contraste y la resolución de las imágenes. Ahora, un nuevo modelo computacional, basado en una arquitectura avanzada de aprendizaje profundo, ofrece tiempos de procesamiento más rápidos y, al mismo tiempo, alcanza o supera la calidad de imagen de los métodos tradicionales.
El modelo, llamado Multi-Stage Residual-BCR Net (m-rBCR), fue desarrollado específicamente para imágenes de microscopía por investigadores del Helmholtz-Zentrum Dresden-Rossendorf (HZDR, Dresde, Alemania) y el Centro Max Delbrück de Medicina Molecular (Berlín, Alemania). Introduce un enfoque novedoso para el procesamiento de imágenes mediante la deconvolución, un método destinado a mejorar el contraste y la resolución de las imágenes digitales capturadas por microscopios ópticos, incluidos los de campo amplio, confocales o de transmisión. La deconvolución aborda el desenfoque de la imagen, que es un tipo de distorsión causada por el sistema óptico, y se puede realizar de dos formas principales: deconvolución explícita y deconvolución basada en aprendizaje profundo.

Las técnicas de deconvolución explícita se basan en el concepto de la función de dispersión de puntos (PSF), que describe cómo la luz procedente de una fuente puntual se dispersa por el sistema óptico, creando un patrón de difracción tridimensional. Esta dispersión hace que la luz desenfocada contribuya a la borrosidad de una imagen grabada. Al conocer la PSF de un sistema, la borrosidad se puede eliminar matemáticamente, lo que produce una representación más clara de la imagen original. Sin embargo, la deconvolución basada en la PSF está limitada por la dificultad de obtener datos de PSF precisos o exactos para ciertos sistemas. Se han desarrollado métodos de deconvolución a ciegas, en los que la PSF se estima a partir de la propia imagen, pero aún presentan desafíos significativos y han logrado avances limitados.
Para abordar esta cuestión, el equipo de investigación ha aplicado técnicas de "resolución de problemas inversos", que han demostrado ser eficaces en microscopía. Los problemas inversos implican determinar los factores subyacentes que conducen a ciertos resultados observados. La resolución de estos problemas normalmente requiere grandes cantidades de datos y algoritmos avanzados de aprendizaje profundo. Al igual que la deconvolución explícita, el objetivo es lograr imágenes de mayor resolución o mejor calidad. Para su enfoque, presentado en el ECCV, el equipo utilizó una red neuronal basada en la física llamada m-rBCR. En el procesamiento de imágenes, existen dos enfoques básicos: trabajar con la representación espacial de una imagen o su representación de frecuencia, la última de las cuales requiere transformar los datos espaciales. Cada método tiene sus ventajas, y la mayoría de los modelos de aprendizaje profundo operan en el dominio espacial, lo que funciona bien para fotografías generales. Sin embargo, las imágenes de microscopía, en particular las de microscopía de fluorescencia, a menudo son monocromáticas y suelen presentar fuentes de luz específicas contra un fondo oscuro.
Para abordar los desafíos únicos de las imágenes de microscopía, m-rBCR comienza con la representación de frecuencia. Este enfoque permite representaciones de datos ópticos más significativas y permite que el modelo resuelva la tarea de deconvolución con muchos menos parámetros en comparación con otros modelos de aprendizaje profundo. El equipo validó el modelo m-rBCR en cuatro conjuntos de datos diferentes: dos conjuntos de datos de microscopía simulados y dos reales. Demostró un alto rendimiento con significativamente menos parámetros de entrenamiento y tiempos de procesamiento más rápidos que los modelos de aprendizaje profundo actuales, al mismo tiempo que superó a los métodos de deconvolución explícitos.
“Esta nueva arquitectura aprovecha una forma olvidada de aprender representaciones más allá de los enfoques clásicos de redes neuronales convolucionales”, dijo el coautor, el profesor Misha Kudryashev, líder del grupo “Biología estructural in situ” del Centro Max Delbrück de Medicina Molecular. “Nuestro modelo reduce significativamente los parámetros potencialmente redundantes. Como muestran los resultados, esto no va acompañado de una pérdida de rendimiento. El modelo es explícitamente adecuado para imágenes de microscopía y, al tener una arquitectura liviana, desafía la tendencia de modelos cada vez más grandes que requieren cada vez más potencia de procesamiento”.