Validan clínicamente prueba de ADN tumoral circulante
Actualizado el 30 May 2022
La secuenciación de próxima generación del ADN tumoral circulante presenta un enfoque prometedor para el diagnóstico del cáncer, que complementa las pruebas de diagnóstico convencionales basadas en tejidos al permitir pruebas en serie mínimamente invasivas y una amplia cobertura genómica a través de una simple extracción de sangre para maximizar el beneficio terapéutico para los pacientes.
El diagnóstico y la subclasificación de los cánceres, fundamentales en cualquier algoritmo de tratamiento del cáncer, se ha logrado históricamente a través del estudio del tejido tumoral primario. La disponibilidad de material de diagnóstico de alta calidad adecuado para tecnologías genómicas de alto nivel, incluida la genotipificación basada en tejidos multiplexados, a menudo es limitante y está asociada con altos riesgos y costos.
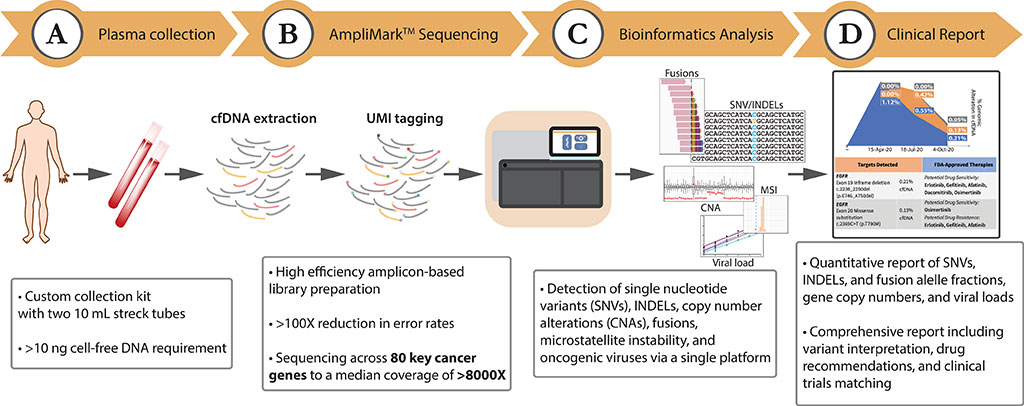
Los científicos de Lucence Diagnostics Pte Ltd (Singapur, Singapur) recolectaron 1.592 muestras clínicas consecutivas incluidas en los análisis observacionales que se recibieron como muestras clínicas de rutina y que se sometieron a un procesamiento e informe estándar en un laboratorio acreditado por CAP y CLIA. Se recolectaron tubos de sangre total para cada paciente con el Streck Cell-Free DNA BCT (Streck, La Vista, NE, EUA), se enviaron a Lucence Diagnostics a temperatura ambiente y se procesaron dentro de las 6 a 48 horas posteriores a la recolección de la muestra.
El equipo utilizó la prueba LiquidHALLMARK de Lucence Diagnostics, que es un ensayo de secuenciación de próxima generación, basado en amplicones, desarrollado para el perfil genómico del ADN libre de células derivado de plasma (ADNlc). El panel integral de 80 genes perfila mutaciones puntuales, inserciones/eliminaciones, alteraciones del número de copias y fusiones de genes, y además detecta virus oncogénicos (virus de Epstein-Barr (VEB) y virus de la hepatitis B (VHB)) e inestabilidad de microsatélites (MSI).
Los investigadores informaron que la validación analítica utilizando materiales genéticos de referencia demostró una sensibilidad del 99,4 % para mutaciones puntuales y del 95,8 % para inserciones/deleciones al 0,1 % de frecuencia alélica variante (VAF), y una sensibilidad del 91,7 % para fusiones de genes al 0,5 % de VAF. En muestras no cancerosas, se observó una alta especificidad (≥99,9999 % por base).
También se determinó empíricamente el límite de detección para alteraciones del número de copias, VEB, VHB y la MSI. La comparación ortogonal de células variantes del receptor del factor de crecimiento epidérmico (EGFR) realizadas por LiquidHALLMARK y un método de reacción en cadena de la polimerasa específico de alelo (AS-PCR) de referencia para 355 muestras de cáncer de pulmón reveló una concordancia general del 93,8 %, mientras que la validación externa con cobas la Prueba de Mutación del EGFR vobas v2 (Roche, Basilea, Suiza) para 50 muestras de cáncer de pulmón demostró una concordancia general del 84,0 %, con una tasa de concordancia del 100 % para las variantes de EGFR por encima del 0,4 % de VAF.
La aplicación clínica de LiquidHALLMARK en 1.592 pacientes consecutivos demostró una alta tasa de detección (74,8 % de ADN tumoral circulante (ADNtc) positivo en muestras de cáncer) y una amplia capacidad de acción (50,0 % de las muestras de cáncer que albergan alteraciones con evidencia biológica de capacidad de acción). Entre los cánceres de pulmón positivos para ADNtc, el 72,5 % albergaba al menos un biomarcador con una indicación de fármaco aprobada por las guías.
Los autores concluyeron que sus resultados establecieron la alta sensibilidad, especificidad, exactitud y precisión del ensayo LiquidHALLMARK y respaldan su aplicación clínica para pruebas genómicas en sangre. El estudio fue publicado el 29 de abril de 2022 en la revista PLOS ONE .
Enlaces relacionados:
Lucence Diagnostics Pte Ltd
Streck
Roche