Análisis en una sola célula descubre un programa regulador en una leucemia rara
Por el equipo editorial de LabMedica en español
Actualizado el 25 Dec 2019
La identificación de las causas de las enfermedades humanas requiere la desconvolución de los fenotipos moleculares anormales que abarcan la accesibilidad del ADN, la expresión de genes y la abundancia de las proteínas. La leucemia aguda de fenotipo mixto presenta características tanto de leucemia mieloide aguda como de leucemia linfoide aguda y, como tal, está marcada por características de linajes hematopoyéticos múltiples.Actualizado el 25 Dec 2019
La leucemia aguda de fenotipo mixto es un tipo de leucemia muy raro en el que se produce más de un tipo de leucemia al mismo tiempo. Esto puede suceder cuando una persona tiene: blastos de leucemia linfoide aguda (LLA) (células cancerosas) y blastos de leucemia mieloide aguda (LMA) al mismo tiempo o blastos leucémicos que tienen características de LLA y de LMA en la misma célula.
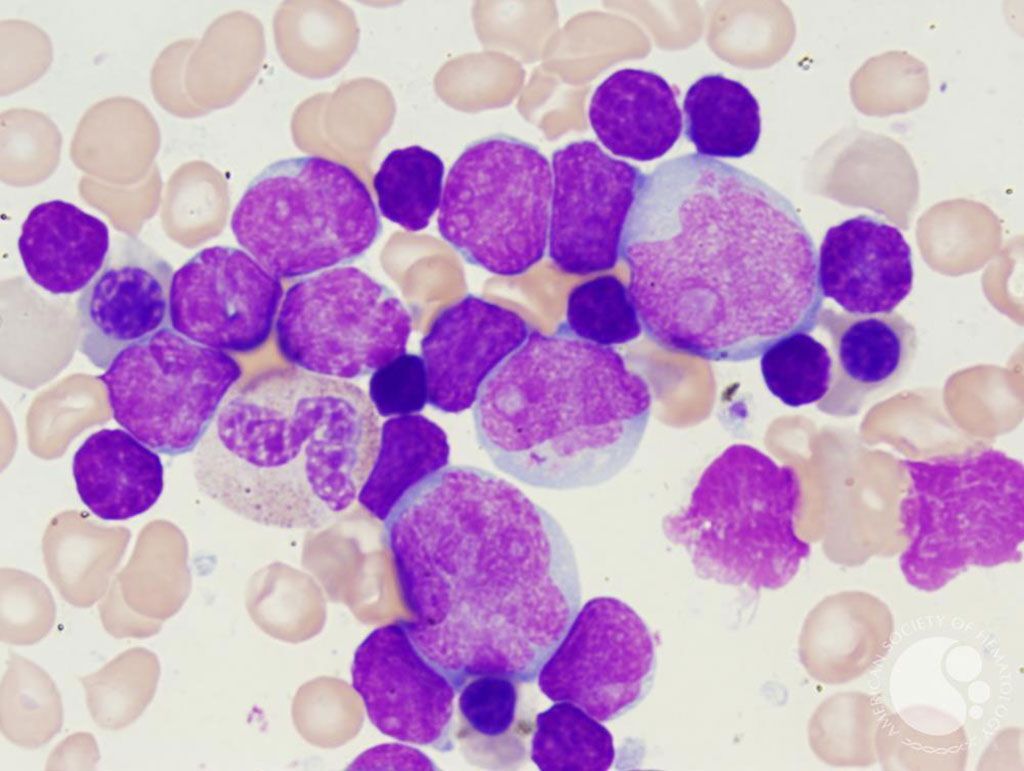
Imagen: Frotis de médula ósea de un paciente con leucemia aguda de fenotipo mixto. El frotis de aspirado de médula tiene 71% de blastos por recuento diferencial, con una morfología dimórfica similar a la de la sangre periférica con numerosos blastos con una morfología dimórfica (Fotografía cortesía de Elizabeth Courville, MD).
Los científicos de la Facultad de Medicina de la Universidad de Stanford (Stanford, CA, EUA) identificaron las características moleculares patológicas de la leucemia aguda de fenotipo mixto al analizar primero los perfiles transcriptómicos y epigenéticos unicelulares de las células sanguíneas sanas durante su desarrollo. Una vez que establecieron los perfiles de esas células sanas, examinaron cómo se comparaban los perfiles de las células leucémicas.
El equipo realizó una indexación celular basada en gotitas de transcriptomos y epítopos mediante secuenciación (CITE-seq) y un ensayo unicelular para cromatina accesible por transposasa mediante secuenciación (scATAC-seq) de más de 35.000 células sanas de médula ósea y de sangre periférica. Con esto, generaron mapas multiómicos de hematopoyesis. Validaron los mapas y descubrieron que reflejaban las características fenotípicas, transcriptómicas y epigenéticas esenciales del desarrollo sanguíneo. Desarrollaron un marco para analizar las firmas del desarrollo hematopoyético a nivel de las células individuales. Con esto, buscaron examinar cómo esas firmas diferían entre las células sanas y las leucémicas.
El equipo analizó miles de células individuales de muestras de leucemia aguda de fenotipo mixto (MPAL) utilizando tanto CITE-seq como scATAC-seq e identificó 4.616 genes que estaban regulados por incremento diferencial y 72.196 picos significativamente regulados. Proyectaron estos análisis unicelulares en sus mapas hematopoyéticos para encontrar la diversidad de expresión epigenética y génica y agruparon las células en amplios compartimentos de desarrollo hematopoyético. Se concentraron en los factores de transcripción que podrían regular estos programas de leucemia y descubrieron que los motivos RUNX1 se enriquecían entre ciertos subconjuntos de MPAL.
RUNX1, señalaron, es un gen frecuentemente mutado en las neoplasias hematológicas, y descubrieron 732 genes regulados por un elemento distal que contiene RUNX1 en al menos dos subconjuntos de MPAL. Además, el CD69 que se ha relacionado con la activación de linfocitos a través de la señalización JAK-STAT y la retención de linfocitos en los órganos linfoides se reguló de forma diferencial en casi todos los subconjuntos de MPAL. Los autores concluyeron que sus resultados demuestran cómo el análisis integrativo y multiómico de células individuales en el marco del desarrollo normal, puede revelar mecanismos moleculares de enfermedad tanto únicos como compartidos a partir de muestras de pacientes. El estudio fue publicado el 2 de diciembre de 2019 en la revista Nature Biotechnology.
Enlace relacionado:
Facultad de Medicina de la Universidad de Stanford