Biopsias líquidas detectan los tumores óseos pediátricos con base en los perfiles epigenéticos
Por el equipo editorial de LabMedica en español
Actualizado el 16 Jun 2021
La secuenciación de ADN libre de células (ADNlc) en la sangre de pacientes con cáncer (biopsia líquida) brinda oportunidades atractivas para el diagnóstico temprano, la evaluación de la respuesta al tratamiento y el monitoreo de enfermedades mínimamente invasivas. Como consecuencia, el análisis de ADNlc es muy prometedor para la oncología de precisión y las terapias personalizadas y actualmente se evalúa en una amplia gama de estudios clínicos.Actualizado el 16 Jun 2021
El sarcoma de Ewing (SEw) constituye un cáncer modelo ideal para establecer y validar el análisis de biopsia líquida basada en fragmentos para tumores pediátricos. El SEw tiene una firma epigenética única con asociaciones clínicas establecidas, lo que constituye un marcador epigenético potencial para el diagnóstico temprano y la clasificación de tumores con base en el ADNlc. Además, los SEw tienen aberraciones genéticas bien establecidas que se pueden usar para comparar.

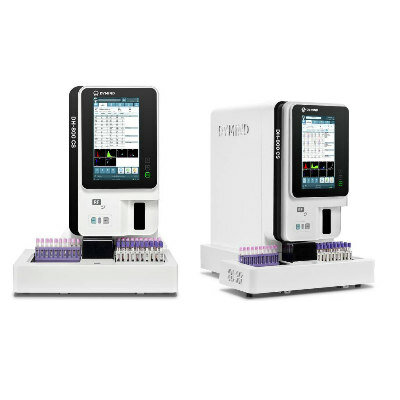
Imagen: El QIAsymphony SP permite la preparación de muestras de ADN, ARN y ácidos nucleicos bacterianos y virales a partir de una amplia gama de materiales de partida (Fotografía cortesía de Qiagen)
Un equipo internacional dirigido por científicos del Instituto de Investigación del Cáncer Infantil St. Anna (Viena, Austria) y sus colegas, incluyó 200 muestras de plasma de 95 pacientes con SEw y 41 muestras de plasma de 31 pacientes con otros tipos de sarcoma: sarcoma de tipo SEw (tres pacientes, dos de los cuales fueron positivos para el gen de fusión CIC-DUX4), osteosarcoma (ocho pacientes), rabdomiosarcoma (12 pacientes), sarcoma sinovial (tres pacientes) y otros tipos de sarcoma (cinco pacientes). Se utilizaron como controles muestras de plasma de 22 individuos sanos (de 24 a 50 años).
El ADNlc se aisló usando el kit de ADN circulante QIAsymphony con el instrumento QIAsymphony SP o el kit cfDNA QIAampMinElute (Qiagen, Hilden, Alemania). Se generaron bibliotecas de secuenciación de genoma completo a partir de 10 ng de ADNlc. Las bibliotecas finales se eluyeron en 20 µl de agua libre de nucleasas, cuantificadas con el kit de ensayo Qubit dsDNA HS (Thermo Fisher Scientific, Waltham, MA, EUA). Los ensayos específicos del paciente para la detección y cuantificación de genes de fusión se diseñaron siguiendo las pautas de Bio-Rad ddPCR (Bio-Rad, Hércules, CA, EUA).
El equipo informó que, después de confirmar que podían detectar ADN derivado de tumores en sus muestras de ADN libre de células, notaron que el ADN derivado de tumores tendía a ser más corto que el ADN libre de células que se encuentra entre los controles sanos. Estos fragmentos a menudo tenían un tamaño de aproximadamente 167 pares de bases, lo que corresponde a la longitud del ADN que envuelve un nucleosoma más el ADN enlazador. Estos fragmentos de ADN derivados de tumores más cortos se encontraron además entre pacientes en los que no se detectaron cambios genéticos. Esto sugirió que se podrían usar los patrones de fragmentación para identificar el ADN derivado de tumores independientemente de las alteraciones genéticas.
Los científicos desarrollaron aún más una herramienta que denominaron LIQUORICE, para: estimación de cobertura de regiones de interés de biopsia líquida, que superpone estos perfiles de fragmentos de ADN libres de células en todo el genoma sobre un conjunto predeterminado de regiones genómicas donde, frecuentemente, ocurren los cambios epigenéticos según el tipo de cáncer. De esa manera, desarrolla una firma de consenso de cobertura de fragmentos en esas regiones. Además, desarrollaron clasificadores de aprendizaje automático para no solo diferenciar a los individuos con cáncer de los controles, sino también entre diferentes tipos de sarcoma pediátrico, sin depender de los cambios genéticos recurrentes. Cuando probaron sus clasificadores, encontraron que eran muy sensibles.
Los autores concluyeron que su estudio demostró cómo la secuenciación profunda del genoma completo de ADNlc permite la detección, clasificación y monitoreo completos de tumores pediátricos con base en sus perfiles genéticos y epigenéticos, proporcionando así un método clínicamente relevante para el análisis de biopsia líquida en cánceres con pocas o ninguna alteración genética. El estudio fue publicado el 28 de mayo de 2021 en la revista Nature Communications.
Enlace relacionado:
Instituto de Investigación del Cáncer Infantil St. Anna
Thermo Fisher Scientific
Bio-Rad